Система управления сайтами DIAFAN.CMS
Разрабатывайте, управляйте,
продвигайте
DIAFAN.CMS – универсальная система.
Идеально подходит большинству веб-студий для разработки и
поддержки клиентских решений.
Демо Купить лицензию Стать партнёром
Будет удобно
Админка DIAFAN.CMS – одна из самых простых и интуитивно понятных. Разберётся даже ребёнок.
Адаптивность
Управлять сайтом одинаково комфортно с любого устройства
Настраиваемый интерфейс
Пункты админки можно настроить как угодно: оставить только то, что не навредит юзабилити
Edit in place
Пользовательское редактирование прямо на страницах сайта, в формате «что вижу, то и правлю»
Drag&Drop
Простое перетаскивание элементов в списке страниц, меню, каталоге
На виду только важное
Интерфейс не перегружен непонятными новичку функциями – редкое и сложное спрятано от глаз рядового пользователя
Простая настройка
Все возможности доступны из админки, без подключения к FTP
Мультиязычность
Панель управления на русском или английском, публичная часть сайта – на любом языке
Лучшие для SEO По версии портала SEONEWS
Всё для продвижения и оптимизации: гибкая настройка ЧПУ, мета-тегов, корректная отдача заголовков last-modify и служебных страниц с кодами 404, 403, 301, 503. Карта сайта и sitemap.xml создаются автоматически.
Карта сайта и sitemap.xml создаются автоматически.
Производительные и нагрузоустойчивые
Лидер в многочисленных тестах на нагрузки. На простом виртуальном хостинге система выдерживает сотни одновременных посетителей не создавая нагрузки на сервер и не заставляя никого ждать. Подробнее об исследованиях >>>
Море возможностей
Модули
Более 30 пакетов готовых решений: новости, магазин, статьи, фотогалереи, опросы, рассылки, форумы, объявления, файловые архивы, комментарии и многое другое. Любой модуль можно доработать, изменить, расширить. А можно написать свой.
Динамические блоки и модуль Контент-конструктор
Позволяют получить любое количество произвольного контента на любой странице сайта.
Интернет-магазин
Модуль, который содержит практически все, что нужно для торговли в сети: онлайн-платежи, дополнительные характеристики товаров, импорт/экспорт и многое другое.
Интеграция с 1С
Полная и официальная совместимость с «1С: Предприятие» на уровне обмена данными. Можно автоматически загружать на сайт товары из «1С», а также выгружать заказы с сайта обратно в программу.
Можно автоматически загружать на сайт товары из «1С», а также выгружать заказы с сайта обратно в программу.
Дополнения
Дополнительные модули и решения, а также шаблоны дизайна – как тематические, так и универсальные.
Автоматические обновления
Бесплатны в течение года после активации лицензии, проходят в полуавтоматическом режиме. Система распознает измененные и доработанные веб-мастером файлы и выносит их в отдельную папку.
Партнерская программа
Персональные скидки на покупку лицензий, бесплатные лицензии для тестовых и служебных сайтов, оперативная коммерческая поддержка.
Полезные сервисы
Документация
От А до Я. Есть все: разделы для веб-мастеров и верстальщиков, разбор типовых проблем, ответы на распространенные вопросы, а также вебинары, видеоуроки и архивная документация.
Обучение
Учись у лучших – освой платформу в совершенстве. Любой формат: от базовых видео-лекций до углубленных занятий с преподавателем. Получи сертификат, разрабатывай собственные модули и зарабатывай на них.
Получи сертификат, разрабатывай собственные модули и зарабатывай на них.Биржа исполнителей PRO.USER
Получи заказ на разработку или найди подрядчика. Здесь все свои и все по-честному: только проверенные заказчики и исполнители.
Отправка СМС из DIAFAN.CMS
1.Описание
С помощью дополнения «TargetSMS.ru: СМС-рассылки и уведомления», реализованного совместно с DIAFAN, Вы можете отправлять массовые СМС-рассылки и настраивать автоматические СМС-уведомления по заданным событиям администратору и покупателям интернет-магазина, работающего на платформе DIAFAN.CMS.
2.Функционал
- Регистрация Вашего имени отправителя бесплатно.
- Возможность отправки СМС-уведомлений администратора интернет-магазина о поступлении новых заказов.

- Отправка СМС из модулей «Обратная связь», «Оформление заказа», «Комментарии», «Вопрос-Ответ», «Рассылки» (Подключать SMS-уведомления нужно в настройках соответствующего модуля).
ВАЖНО: если Вам нужна персонализация текстов СМС-сообщений, то это можно сделать при помощи других модулей интеграции.
3.Установка дополнения
- Пройдите регистрацию в нашем сервисе СМС-рассылок. После регистрации Вы получите логин, пароль, тестовое имя отправителя и тестовый баланс для проверки работы сервиса СМС-рассылок
- Установите наше дополнение «TargetSMS.ru: СМС-рассылки и уведомления» одним из описанных ниже способов.
3.1.Способ 1. Установка дополнения из маркетплейса DIAFAN.CMS
- Зайдите в административную панель Вашего интернет-магазина, работающего на платформе DIAFAN.CMS
- Зайдите в пункт меню «Сервис» — «Дополнения для CMS»
- Нажмите на кнопку «Каталог дополнений», найдите в каталоге наше приложение, выделите его галочкой и нажмите «Установить на сайте»
3.
 2.Способ 2. Установка дополнения вручную
2.Способ 2. Установка дополнения вручную- Зайдите на страницу нашего дополнения в маркетплейсе DIAFAN.CMS и нажмите на кнопку «Скачать»
- Далее необходимо вручную создать тему и загрузить в неё архив с дополнением (раздел меню «Темы и дизайн» > «Добавить вручную») либо загрузить скаченный архив в уже установленную тему. Можно также файлы скаченного архива вручную (аккуратно) перенести в файловую систему Вашего интернет-магазина в соответствующие места.
4.Активация дополнения
- Зайдите в пункт меню «Настройки» — «Параметры сайта»
- В низу формы настроек поставьте галочку в пункте «Подключить SMS-уведомления» и заполните форму, указав полученные при регистрации в нашем сервисе логин, пароль и согласованное или тестовое имя отправителя (Внимание: не указывайте произвольные/не согласованные с нами имена отправителей — сервис работает только с заранее прописанными на нашей стороне/согласованными именами отправителей). Нажмите кнопку «Сохранить».

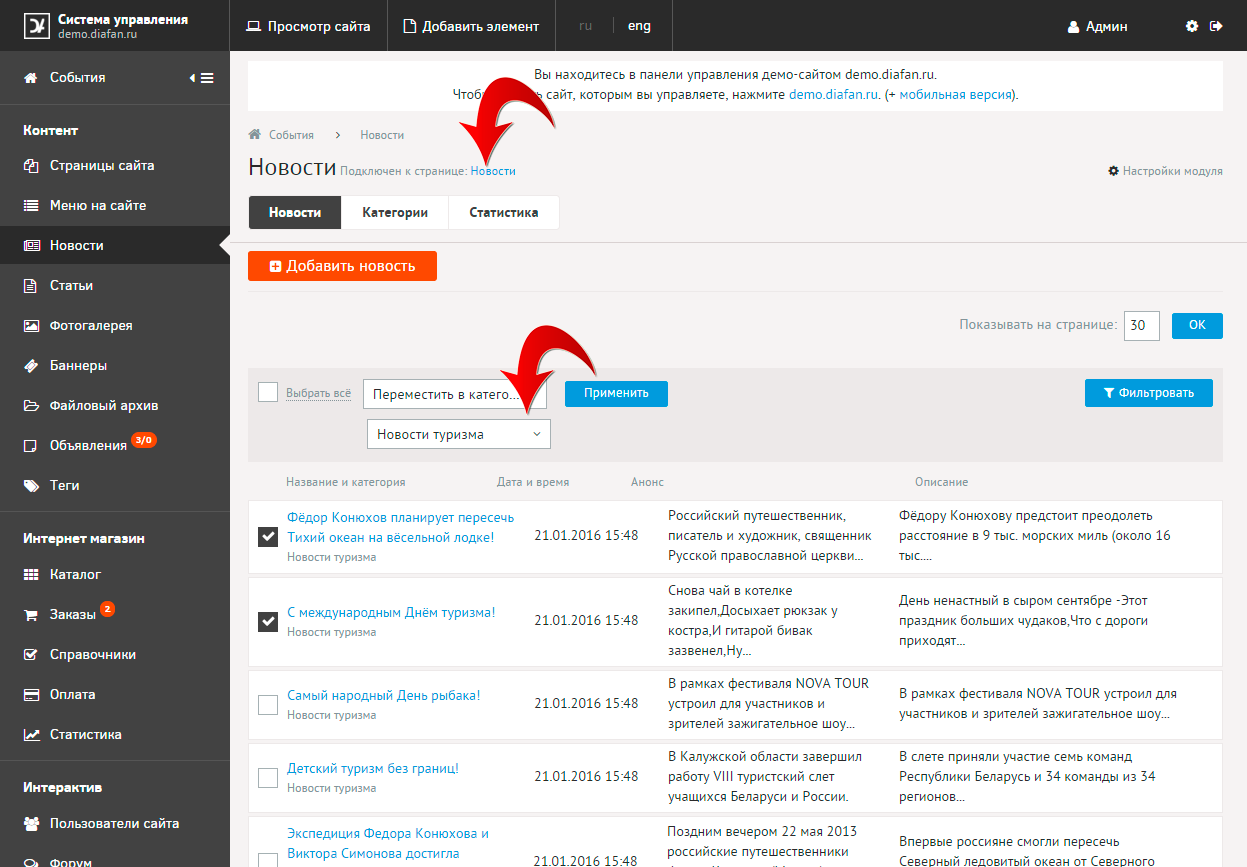
5.СМС-уведомление о поступлении нового заказа администратору интернет-магазина
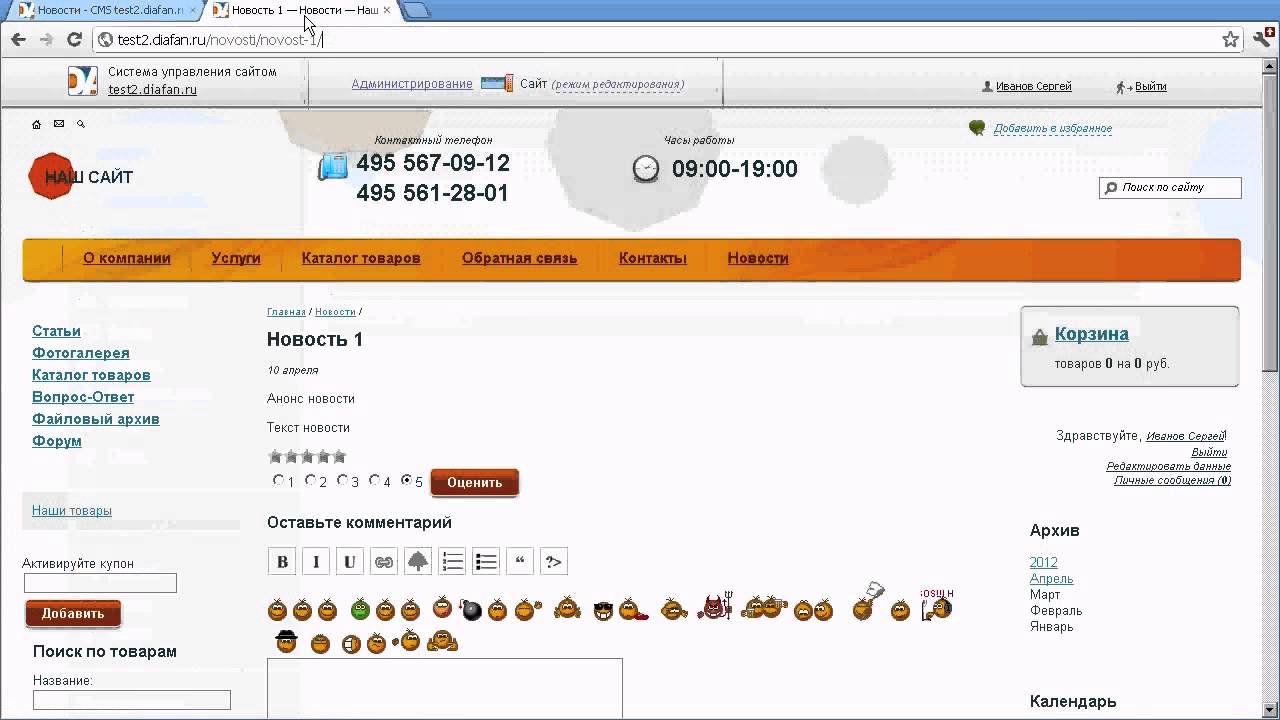
- Пройдите в меню «Интернет магазин» — «Заказы»
- Нажмите на звездочку «Настройки модуля» и откройте вкладку «Сообщения и уведомления»
- В форме настроек модуля поставьте галочку «Уведомлять о поступлении новых заказов по SMS», внесите номер телефона администратора интернет-магазина в формате +79001234567 и введите текст СМС.
6.СМС-уведомление о поступлении нового вопроса в модуле «Вопрос-Ответ»
- Пройдите в меню «Интерактив» — «Вопрос-Ответ»
- Нажмите на звездочку «Настройки модуля» и откройте вкладку «Сообщения и уведомления»
- В форме настроек модуля поставьте галочку «Уведомлять о поступлении новых вопросов по SMS», внестите номер телефона администратора интернет-мазазина в формате +79001234567 и введите текст СМС.
7.СМС-уведомление о поступлении нового вопроса в модуле «Обратная связь»
- Пройдите в меню «Интерактив» — «Обратная связь»
- Нажмите на звездочку «Настройки модуля» и откройте вкладку «Сообщения и уведомления»
- В форме настроек модуля поставьте галочку «Уведомлять о поступлении новых сообщений по SMS», внестите номер телефона администратора интернет-мазазина в формате +79001234567 и введите текст СМС.

8.СМС-уведомление о поступлении нового комментария в модуле «Комментарии»
- Пройдите в меню «Интерактив» — «Комментарии»
- Нажмите на звездочку «Настройки модуля» и откройте вкладку «Сообщения и уведомления»
- В форме настроек модуля поставьте галочку «Уведомлять о поступлении новых комментариев по SMS», внестите номер телефона администратора интернет-мазазина в формате +79001234567 и введите текст СМС.
9.Отправка массовой СМС-рассылки из модуля «Рассылки»
- Пройдите в пункт меню «Интераактив» — «Рассылки», откройте вкладку «SMS-рассылки» и нажмите на кнопку «Добавить SMS-рассылку»
- Заполните форму новой СМС-рассылки, указав название (не будет отправлено получателям) и текст СМС. Подпись отправителя будет автоматически взята из настроек, указанных при активации дополнения. Рассылка будет отправлена всем клиентам, осуществившим заказ в Вашем интернет-магазине (все имеющиеся номера телефонов получателей можно увидеть на вкладке «Номера телефонов».
 Там же можно добавить, удалить или дезактивировать номера телефонов). ВАЖНО: все номера телефонов должны быть в международном формате вида +79001234567.
Там же можно добавить, удалить или дезактивировать номера телефонов). ВАЖНО: все номера телефонов должны быть в международном формате вида +79001234567.
РЕКОМЕНДАЦИЯ: заносить номера телефонов в карточки клиентов исключительно в международном формате +79000000001
10.Типовые проблемы и сложности
Самые популярные проблемы и вопросы по работе с интеграцией описаны тут.
Начать рассылку
Тестовые сообщения бесплатно
Титан CMS
Основное содержаниеДаже самое сложное программное обеспечение для управления веб-контентом не поможет вам достичь ваших цифровых целей, если вы не знаете, как лучше всего его использовать. Вот почему команда , стоящая за вашим инструментом , так же важна, как и сам инструмент.
Выбирая Titan CMS, вы получаете прямой доступ к цифровым экспертам, которые создали ее, управляют ею и постоянно совершенствуют ее. Мы узнаем вас и ваш бизнес, поэтому наша команда 
Наша команда расширяет возможности вашей команды
С Titan CMS у вас будет доступ не только к отличному инструменту управления корпоративным контентом, но и к целой команде цифровых экспертов и идейных лидеров — от стратегов до разработчиков, менеджеров по поддержке и обучению, экспертов по маркетингу и аналитике и других специалистов. – все сосредоточено на вашем успехе.
О нас
Специальное обслуживание и поддержка
Клиенты Titan CMS ценят наше исключительное обслуживание клиентов, постоянную поддержку и индивидуальное обучение. У каждого клиента есть специальный менеджер по работе с клиентами, который всегда готов помочь и стремится помочь вам в достижении ваших целей. С Titan CMS вы никогда не одиноки.
Читайте истории клиентов
100% внутренние … Все
Мы не отдаем на аутсорсинг внедрение, разработку, обслуживание, обучение или поддержку. Ничего. Это означает, что вам не придется полагаться на еще одного партнера, чтобы заставить Titan CMS работать на вас (и поддерживать ее работу).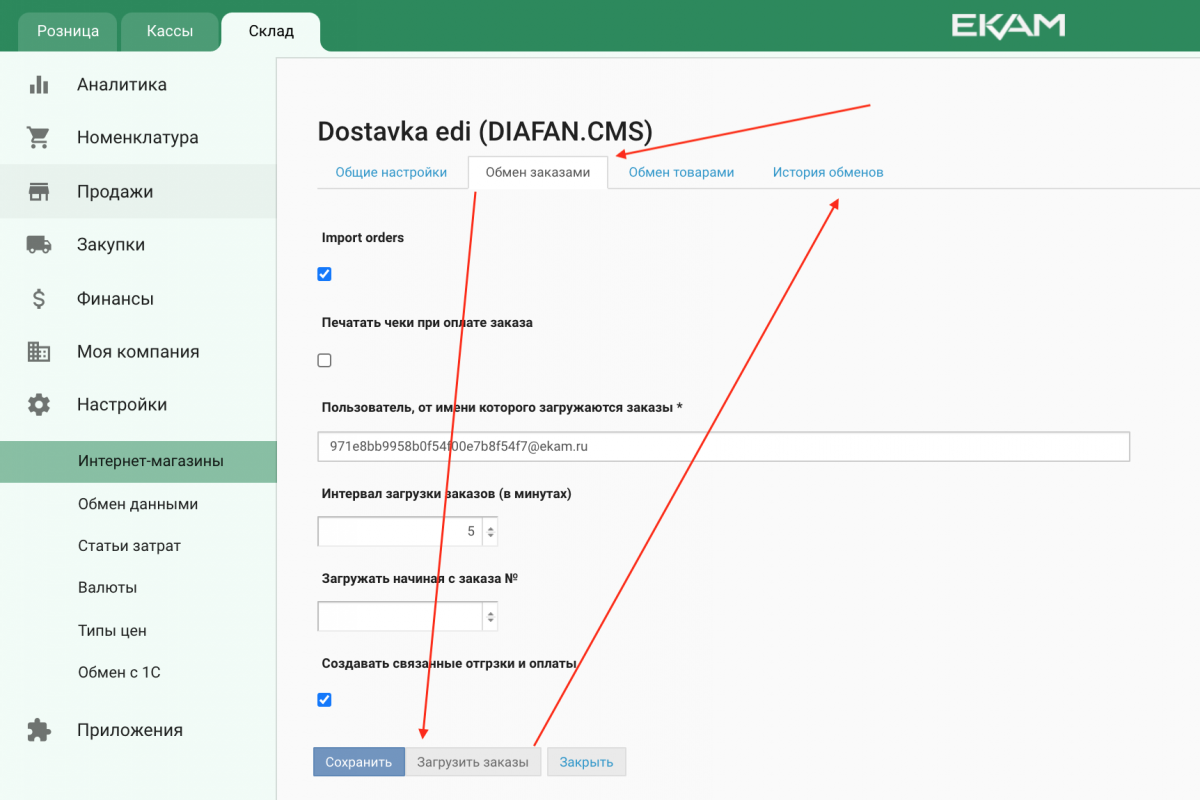 Вместо этого вы приступите к делу в первый же день — с нашей командой прямо на вашей стороне.
Вместо этого вы приступите к делу в первый же день — с нашей командой прямо на вашей стороне.
Узнать больше
Простое в использовании, мощное программное обеспечение CMS
Titan CMS предлагает простой в использовании, интуитивно понятный интерфейс администратора, который оценят как новички, так и специалисты. Благодаря функциям перетаскивания, мощному управлению контентом и данными, безопасности, интеллектуальному поиску по сайту, редактированию WYSIWYG, интеграции со сторонними организациями и многому другому.
См. основные характеристики
Гибкость для удовлетворения ваших потребностей и бюджета
Наша единовременная плата за лицензию дает вам доступ к надежным и доступным функциональным возможностям CMS. Используйте надстройки Titan CMS для наложения различных функций и функций, чтобы вы получали только то, что вам нужно, не платя за то, что вам не нужно.
Просмотреть варианты лицензирования
Отраслевая информация всегда у вас под рукой
Наши цифровые эксперты делятся своим мнением о последних новостях и тенденциях в мире систем управления контентом, веб-разработки и многого другого, а также дают советы по работе с Titan CMS. , приемы и инструкции.
, приемы и инструкции.
Посетите наш блог
Пожалуйста, подождите, пока мы соберем ваши результаты.
Отзывы
Джим Рэнд
«Мы выбрали Northwoods и Titan CMS, потому что нам нужна была интуитивно понятная и простая в использовании CMS-система, которая также не попадала бы в массовые взломы бесплатных платформ или платформ с открытым исходным кодом, таких как Drupal. и Word Press. Нам также нужно было агентство, которое могло бы помочь нам с любыми нашими потребностями в веб-разработке. Titan CMS и Northwoods идеально соответствуют этим требованиям. Их команда разработчиков является первоклассной, и они являются настоящим партнером».
– Джим Рэнд, Pactiv Evergreen
Джефф Ксиазек
«В CelticMKE мы управляем несколькими веб-сайтами на CMS Titan. фильтрации Titan в наши цифровые коллекции Ward Irish Music Archive, которые интегрируются со сторонними базами данных. Конечными результатами являются великолепно выглядящие веб-сайты, которые просты в использовании и обслуживании».
– Джефф Ксиазек, CelticMKE
Донна Боттони
«Обновление до Titan CMS 7 значительно повысило нашу производительность. Мало того, что административный интерфейс стал проще в использовании и навигации, загрузка документов стала молниеносной, а безопасное управление документами на портале стало более оптимизированным. Titan CMS дает нам гибкость и эффективность, которые нам нужны, чтобы совмещать множество различных приоритетов и эффективно достигать наших целей».
– Донна Боттони, Three Pillars Senior Living Communities
% Клиенты говорят, что они
Рекомендую
94%
Клиент Northwoods Sat. Опрос
Вероятность клиента
Продлить
100%
TrustRadius
Качество поддержки
9,7/10
G2
Соотношение цены и качества
4,8/5
Capterra
Пожалуйста, подождите пока мы собираем ваши результаты.
Доверяют Тысячи Пользователей
Dispense-Rite
Кузова гоночных автомобилей Five Star
Flexco
Gehl Food and Beverage
Godfrey & Kahn
Kenall Manufacturing
Milwaukee Irish Fest
Pactiv Evergreen
Серверные продукты
United Way of Greater Milwaukee & Waukesha County
Wisconsin Dells Visitor & Convention Bureau
YMCA округа Большой Вокеша
Получить документацию
Клиенты имеют доступ к онлайн-документации и документации в формате PDF, советам и инструкциям по самым последним версиям Titan CMS. Ознакомьтесь с нашей полной библиотекой документации!
Ознакомьтесь с нашей полной библиотекой документации!
Начать
Получить поддержку
Экспертная поддержка Titan CMS — всего лишь телефонный звонок или письмо по электронной почте!
Служба поддержки Titan CMS
9:00-17:00 КТ Пн.-Пт.
Тел: 414-914-9200
Электронная почта: [email protected]
Получить поддержку
Запросить демонстрацию
Вместе с вами мы создадим пакет лицензий, хостинга и поддержки, соответствующий вашим потребностям и бюджету. Запросите демонстрацию, чтобы начать.
Запросить демонстрацию
Воспроизводимость, специфичность и точность относительного количественного определения с использованием независимого от данных сбора данных на основе библиотеки спектров
1.
Muntel J., Xuan Y., Berger S.T., Reiter L., Bachur R., Kentsis A. и Steen H. (2015)Продвижение открытия биомаркеров мочевого белка путем независимого от данных сбора данных на масс-спектрометре с квадрупольной орбитальной ловушкой.
2. Лю Ю., Чен Дж., Сети А., Ли К.К., Чен Л., Коллинз Б., Жиллет Л.К., Воллшайд Б., Чжан Х. и Эберсолд Р. (2014) Гликопротеомный анализ тканей рака предстательной железы по массе SWATH спектрометрия обнаруживает амидазу N-ацилэтаноламиновой кислоты и протеинтирозинкиназу 7 как признаки агрессивности опухоли. Мол. Клетка. протеомика 13, 1753–1768 [бесплатная статья PMC] [PubMed] [Google Scholar]
3. Мальмстром Э., Килсгард О., Хаури С., Смедс Э., Хервальд Х., Мальмстром Л. и Мальмстром Дж. (2016) Крупномасштабное заключение о происхождении белковой ткани в грамположительной сепсисной плазме с использованием количественной направленной протеомики. Нац. коммун. 7, 10261. [Бесплатная статья PMC] [PubMed] [Google Scholar]
4. Анджо С. И., Санта С. и Манадас Б. (2017) SWATH-MS как инструмент для обнаружения биомаркеров: от фундаментальных исследований до клинических применений. протеомика 17 [PubMed] [Google Scholar]
5. Домон Б. и Эберсолд Р. (2010) Варианты и соображения при выборе стратегии количественной протеомики. Нац. Биотехнолог. 28, 710–721 [PubMed] [Google Scholar]
Домон Б. и Эберсолд Р. (2010) Варианты и соображения при выборе стратегии количественной протеомики. Нац. Биотехнолог. 28, 710–721 [PubMed] [Google Scholar]
6. McDonald WH и Yates JR (2002) Протеомика дробовика и открытие биомаркеров. Дис. Маркеры 18, 99–105 [бесплатная статья PMC] [PubMed] [Google Scholar]
7. Sajic T., Liu Y., and Aebersold R. (2015)Использование независимой от данных масс-спектрометрии с высоким разрешением в исследовании белковых биомаркеров: перспективы и клиническое применение. Протеомика клин. заявл. 9, 307–321 [PubMed] [Google Scholar]
8. Михальски А., Кокс Дж. и Манн М. (2011) Более 100 000 поддающихся обнаружению видов пептидов элюируются в ходе однократных прогонов протеомики, но большинство из них недоступны для зависимых от данных ЖХ-МС/МС. Дж. Протеом Рез. 10, 1785–179 гг.3 [PubMed] [Google Scholar]
9.
Табб Д. Л., Вега-Монтото Л., Рудник П. А., Варият А. М., Хэм А.-Дж. Л., Банк Д. М., Килпатрик Л. Э., Биллхаймер Д. Д., Блэкман Р. К., Кардасис Х. Л., Карр С. А., Клаузер К. Р., Яффе Дж. Д., Ковальски К. А., Нойберт Т. А., Ренье Ф. Э., Шиллинг Б., Тегелер Т. Дж., Ван М., Ван П. , Уайтакер Дж. Р., Циммерман Л. Дж., Фишер С. Дж., Гибсон Б. В., Кинсингер К. Р., Месри М., Родригес Х., Штейн С. Э., Темпст П., Паулович А. Г., Либлер Д. К. и Шпигельман К. (2009 г.)) Повторяемость и воспроизводимость протеомной идентификации с помощью жидкостной хроматографии и тандемной масс-спектрометрии. Дж. Протеом Рез. 9, 761–776. [Бесплатная статья PMC] [PubMed] [Google Scholar]
К., Кардасис Х. Л., Карр С. А., Клаузер К. Р., Яффе Дж. Д., Ковальски К. А., Нойберт Т. А., Ренье Ф. Э., Шиллинг Б., Тегелер Т. Дж., Ван М., Ван П. , Уайтакер Дж. Р., Циммерман Л. Дж., Фишер С. Дж., Гибсон Б. В., Кинсингер К. Р., Месри М., Родригес Х., Штейн С. Э., Темпст П., Паулович А. Г., Либлер Д. К. и Шпигельман К. (2009 г.)) Повторяемость и воспроизводимость протеомной идентификации с помощью жидкостной хроматографии и тандемной масс-спектрометрии. Дж. Протеом Рез. 9, 761–776. [Бесплатная статья PMC] [PubMed] [Google Scholar]
10. Лю Х., Садыгов Р. Г. и Йейтс Дж. Р. 3rd. (2004) Модель случайной выборки и оценки относительного содержания белка в протеомике дробовика. Анальный. хим. 76, 4193–4201 [PubMed] [Google Scholar]
11.
Бондаренко П. В., Челиус Д. и Шалер Т. А. (2002) Идентификация и относительный количественный анализ белковых смесей путем ферментативного расщепления с последующей капиллярной обращенно-фазовой жидкостной хроматографией-тандемной масс-спектрометрией. Анальный. хим. 74, 4741–4749[PubMed] [Google Scholar]
Анальный. хим. 74, 4741–4749[PubMed] [Google Scholar]
12. Чжу В., Смит Дж. В. и Хуанг С. М. (2010) Количественная протеомика без меток на основе масс-спектрометрии. Дж. Биомед. Биотехнолог. 2010, 840518. [Бесплатная статья PMC] [PubMed] [Google Scholar]
13. Пикотти П. и Эберсолд Р. (2012) Избранная протеомика, основанная на мониторинге реакций: рабочие процессы, потенциал, подводные камни и будущие направления. Нац. Методы 9, 555–566 [PubMed] [Google Scholar]
14. Эберсолд Р., Бенсимон А., Коллинз Б.К., Людвиг К. и Сабидо Э. (2016) Приложения и разработки в целевой протеомике: от SRM до DIA/SWATH. протеомика 16, 2065–2067 [PubMed] [Google Scholar]
15. Gillet L.C., Navarro P., Tate S., Rost H., Selevsek N., Reiter L., Bonner R., and Aebersold R. (2012) Целевое извлечение данных из спектров МС/МС, полученных путем независимого сбора данных: новая концепция последовательного и точного анализа протеома. Мол. Клетка. протеомика 11, O111 016717 [бесплатная статья PMC] [PubMed] [Google Scholar]
16. Брудерер Р., Бернхардт О.М., Ганди Т., Миладинович С.М., Ченг Л.Ю., Месснер С., Эренбергер Т., Занотелли В., Бутшайд Ю., Эшер К., Витек О., Риннер О. и Рейтер Л. ( 2015) Расширение границ количественного профилирования протеома за счет независимого от данных сбора и применения к обработанным ацетаминофеном трехмерным микротканям печени. Мол. Клетка. протеомика
14, 14:00–14:10 [бесплатная статья PMC] [PubMed] [Google Scholar]
Брудерер Р., Бернхардт О.М., Ганди Т., Миладинович С.М., Ченг Л.Ю., Месснер С., Эренбергер Т., Занотелли В., Бутшайд Ю., Эшер К., Витек О., Риннер О. и Рейтер Л. ( 2015) Расширение границ количественного профилирования протеома за счет независимого от данных сбора и применения к обработанным ацетаминофеном трехмерным микротканям печени. Мол. Клетка. протеомика
14, 14:00–14:10 [бесплатная статья PMC] [PubMed] [Google Scholar]
17. Бильбао А., Варесио Э., Любан Дж., Страмбио-Де-Кастилия К., Хопфгартнер Г., Мюллер М. и Лисачек Ф. (2015) Стратегии обработки и программные решения для независимого от данных сбора данных в масс-спектрометрии. протеомика 15, 964–980 [PubMed] [Google Scholar]
18. Шао В. и Лам Х. (2017) Тандемные масс-спектральные библиотеки пептидов и их роль в исследованиях протеомики. Масс-спектр. Rev. 36, 634–648 [PubMed] [Google Scholar]
19.
Цоу С.С., Автономов Д., Ларсен Б., Тухолска М., Чой Х., Гинграс А.С. и Несвижский А.И. (2015) DIA-Umpire: комплексная вычислительная платформа для независимой от данных протеомики сбора данных. Нац. Методы
12, 258–264, 257 стр. после 264 [бесплатная статья PMC] [PubMed] [Google Scholar]
Нац. Методы
12, 258–264, 257 стр. после 264 [бесплатная статья PMC] [PubMed] [Google Scholar]
20. Брудерер Р., Зондерманн Дж., Цоу С.С., Баррантес-Фрир А., Штадельманн С., Несвижский А.И., Шмидт М., Рейтер Л. и Гомес-Варела Д. (2017) Новые целевые подходы для количественной оценки данных. независимая масс-спектрометрия. протеомика 17 [Бесплатная статья PMC] [PubMed] [Google Scholar]
21. Weisbrod C.R., Eng JK, Hoopmann MR, Baker T. и Bruce J.E. (2012) Точный масс-анализ пептидных фрагментов: мультиплексная идентификация и количественная оценка пептидов. Дж. Протеом Рез. 11, 1621–1632 [бесплатная статья PMC] [PubMed] [Google Scholar]
22. Людвиг К., Джиллет Л., Розенбергер Г., Амон С., Коллинз Б. К. и Эберсолд Р. (2018) SWATH-MS на основе независимого сбора данных для количественной протеомики: учебное пособие. Мол. Сист. биол. 14, е8126. [Бесплатная статья PMC] [PubMed] [Google Scholar]
23.
Венейбл Дж. Д., Донг М. К., Вольшлегель Дж., Диллин А. и Йейтс Дж. Р. (2004) Автоматизированный подход к количественному анализу смесей сложных пептидов по тандемным масс-спектрам. Нац. Методы
1, 39–45 [PubMed] [Google Scholar]
Р. (2004) Автоматизированный подход к количественному анализу смесей сложных пептидов по тандемным масс-спектрам. Нац. Методы
1, 39–45 [PubMed] [Google Scholar]
24. Барковиц К., Линден А., Галоцци С., Шильде Л., Пачарра С., Молленхауэр Б., Стопель Н., Штайнбах С., Мэй К., Ушкорайт Дж., Эйзенахер М. и Маркус К. (2018 г. ) Характеристика спинномозговой жидкости с помощью независимой от данных масс-спектрометрии. Дж. Протеом Рез. 17, 3418–3430 [PubMed] [Google Scholar]
25. Келструп С. Д., Беккер-Дженсен Д. Б., Арри Т. Н., Хогребе А., Хардер А. и Олсен Дж. В. (2018) Оценка производительности Q Exactive HF-X для протеомики дробовика. Дж. Протеом Рез. 17, 727–738 [PubMed] [Google Scholar]
26. Вовинкель Дж., Капуано Ф., Кэмпбелл К., Дири М.Дж., Лилли К.С. и Ралсер М. (2013) Прелесть отсутствия (меток): методы подготовки образцов для SWATH-MS и целевой протеомики следующего поколения. F1000рез 2, 272. [Бесплатная статья PMC] [PubMed] [Google Scholar]
27.
Розенбергер Г. , Кох К.С., Гуо Т., Рост Х.Л., Коувонен П., Коллинз Б.К., Хойзел М., Лю Ю., Карон Э., Вичалковски А., Файни М., Шуберт О.Т., Фариди П., Эбхардт Х.А. , Матондо М., Лам Х., Бадер С.Л., Кэмпбелл Д.С., Дойч Э.В., Мориц Р.Л., Тейт С. и Эберсолд Р. (2014) Хранилище анализов для количественного определения 10 000 белков человека с помощью SWATH-MS. науч. Данные
1, 140031. [Бесплатная статья PMC] [PubMed] [Google Scholar]
, Кох К.С., Гуо Т., Рост Х.Л., Коувонен П., Коллинз Б.К., Хойзел М., Лю Ю., Карон Э., Вичалковски А., Файни М., Шуберт О.Т., Фариди П., Эбхардт Х.А. , Матондо М., Лам Х., Бадер С.Л., Кэмпбелл Д.С., Дойч Э.В., Мориц Р.Л., Тейт С. и Эберсолд Р. (2014) Хранилище анализов для количественного определения 10 000 белков человека с помощью SWATH-MS. науч. Данные
1, 140031. [Бесплатная статья PMC] [PubMed] [Google Scholar]
28. Wu J. X., Song X., Pascovici D., Zaw T., Care N., Krisp C. и Molloy M. P. (2016) Производительность масс-спектрометрии SWATH с использованием расширенных библиотек анализа пептидов MS/MS. Мол. Клетка. Протеомика. 15, 2501–2514 [бесплатная статья PMC] [PubMed] [Google Scholar]
29. Говарт Э., Ван Стендам К., Виллемс С., Воссарт Л., Даэненс М. и Дефорс Д. (2017) Сравнение протеомики фракционирования для создания местной библиотеки SWATH. протеомика 17, 1700052-n/a [бесплатная статья PMC] [PubMed] [Google Scholar]
30.
Гессулат С., Шмидт Т., Золг Д. П. , Самарас П., Шнатбаум К., Зервек Дж., Кнауте Т., Рехенбергер Дж., Деланге Б., Хумер А., Раймер У., Эрлих Х. К., Айше С., Кустер Б. и Вильгельм М. (2019) Prosit: прогнозирование тандемных масс-спектров пептидов с помощью глубокого обучения в масштабах всего протеома. Нац. Методы
16, 509–518 [PubMed] [Google Scholar]
, Самарас П., Шнатбаум К., Зервек Дж., Кнауте Т., Рехенбергер Дж., Деланге Б., Хумер А., Раймер У., Эрлих Х. К., Айше С., Кустер Б. и Вильгельм М. (2019) Prosit: прогнозирование тандемных масс-спектров пептидов с помощью глубокого обучения в масштабах всего протеома. Нац. Методы
16, 509–518 [PubMed] [Google Scholar]
31. Тивари С., Леви Р., Гутенбруннер П., Салинас Сото Ф., Паланиаппан К. К., Деминг Л., Берндл М., Брант А., Симерманчич П. и Кокс Дж. (2019 г.) Высококачественное предсказание спектра МС/МС для зависимого от данных и независимого от данных анализа данных сбора данных. Нац. Методы 16, 519–525 [PubMed] [Google Scholar]
32. Габриэль Р., Мартенс Л. и Дегроев С. (2019) Обновленный веб-сервер MS 2 PIP обеспечивает быстрое и точное прогнозирование пиковой интенсивности MS 2 для нескольких методов фрагментации, инструментов и методов маркировки. Нуклеиновые Кислоты Res. 47, W295–W299 [бесплатная статья PMC] [PubMed] [Google Scholar]
33. Рёст Х. Л., Заксенберг Т., Айше С., Билов К., Вайссер Х., Айхелер Ф., Андреотти С., Эрлих Х.-К., Гутенбруннер П., Кенар Э., Лян Х., Нансен С., Нильс Л., Пфойффер Дж., Розенбергер Г., Рюрик М., Шмитт Ю., Фейт Дж., Вальцер М., Войнар Д., Вольский В. Э., Шиллинг О., Чоудхари Дж. С., Мальмстрем Л., Эберсолд Р., Райнерт К. и Кольбахер О. (2016) OpenMS: гибкая программная платформа с открытым исходным кодом для анализа данных масс-спектрометрии. Нац. Методы
13, 741. [PubMed] [Google Scholar]
Рёст Х. Л., Заксенберг Т., Айше С., Билов К., Вайссер Х., Айхелер Ф., Андреотти С., Эрлих Х.-К., Гутенбруннер П., Кенар Э., Лян Х., Нансен С., Нильс Л., Пфойффер Дж., Розенбергер Г., Рюрик М., Шмитт Ю., Фейт Дж., Вальцер М., Войнар Д., Вольский В. Э., Шиллинг О., Чоудхари Дж. С., Мальмстрем Л., Эберсолд Р., Райнерт К. и Кольбахер О. (2016) OpenMS: гибкая программная платформа с открытым исходным кодом для анализа данных масс-спектрометрии. Нац. Методы
13, 741. [PubMed] [Google Scholar]
34. Пфойффер Дж., Саксенберг Т., Алка О., Вальцер М., Филлбрунн А., Нильсе Л., Шиллинг О., Райнерт К. и Кольбахер О. (2017) OpenMS — платформа для воспроизводимого анализа данных масс-спектрометрии . Дж. Биотехнология. 261, 142–148 [PubMed] [Google Scholar]
35.
Наварро П., Кухарев Дж., Жилле Л. К., Бернхардт О. М., Маклин Б., Рёст Х. Л., Тейт С. А., Цоу К. К., Райтер Л., Дистлер У., Розенбергер Г., Перес-Ривероль Ю., Несвижский А. И., Эберсолд Р. ., and Tenzer S. (2016) Многоцентровое исследование оценивает программные инструменты для количественной оценки протеома без меток.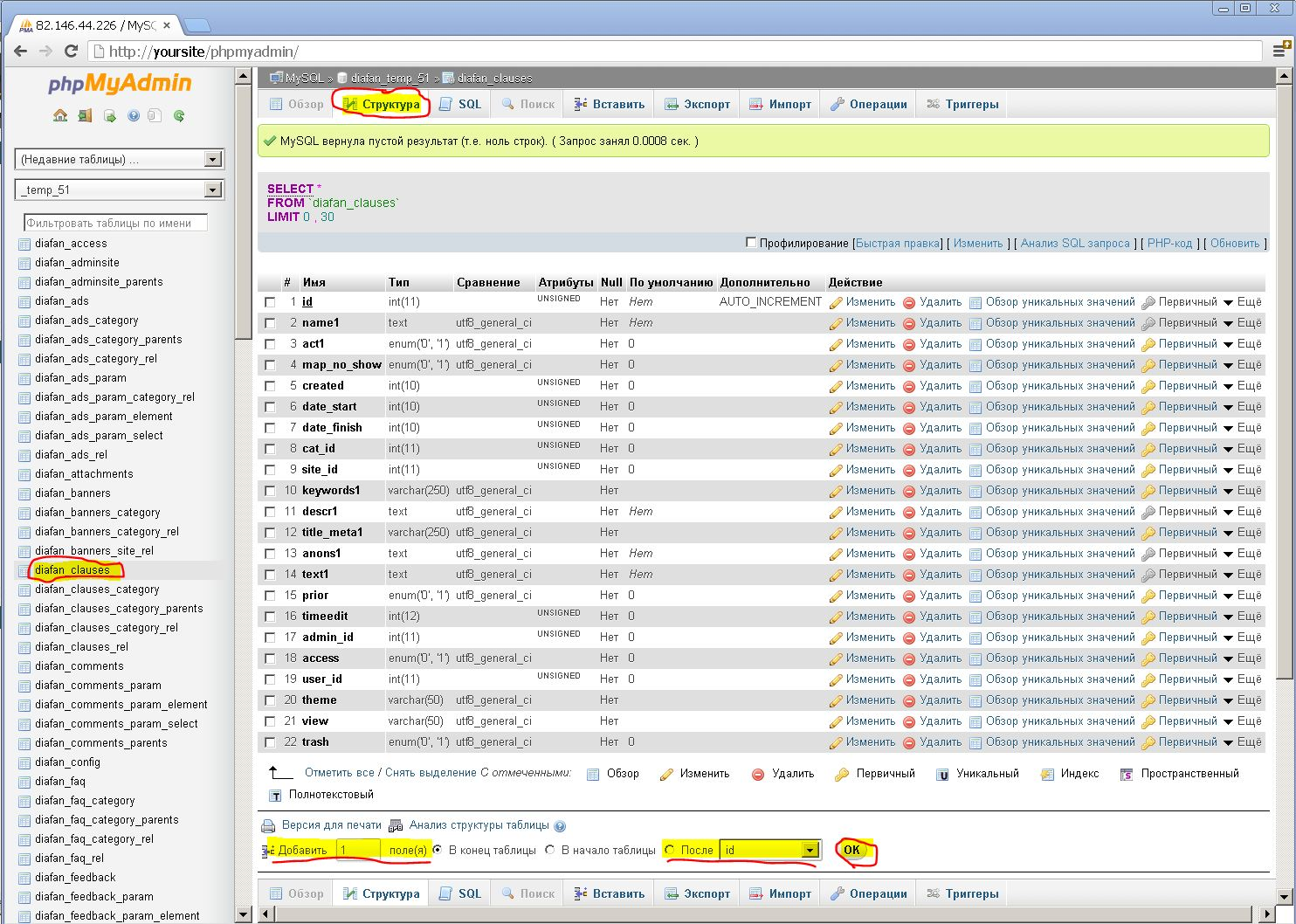 Нац. Биотехнолог. 34, 1130–1136 [бесплатная статья PMC] [PubMed] [Google Scholar]
Нац. Биотехнолог. 34, 1130–1136 [бесплатная статья PMC] [PubMed] [Google Scholar]
36. Рёст Х.Л., Розенбергер Г., Наварро П., Джиллет Л., Миладинович С.М., Шуберт О.Т., Вольски В., Коллинз Б.К., Мальмстрем Дж., Мальмстрем Л. и Эберсолд Р. (2014) OpenSWATH обеспечивает автоматизированный целенаправленный анализ независимое от данных получение данных MS. Нац. Биотехнолог. 32, 219–223 [PubMed] [Google Scholar]
37. Плам С., Хеллинг С., Тейсс К., Лейте Р. Э. П., Мэй К., Якоб-Фильо В., Эйзенахер М., Кульманн К., Мейер Х. Э., Ридерер П., Гринберг Л. Т., Герлах М. и Маркус К. (2013)Комбинированное обогащение гранул нейромеланина и синаптосом из ткани компактной части черной субстанции человека для протеомного анализа. Дж. Протеомика 94, 202–206 [бесплатная статья PMC] [PubMed] [Google Scholar]
38.
Вискаино Дж. А., Чордас А., Дель-Торо Н., Дианес Дж. А., Грисс Дж., Лавидас И., Майер Г., Перес-Риверол Ю., Райзингер Ф., Тернент Т., Сюй К. В., Ван Р. и Hermjakob H. (2016) Обновление базы данных PRIDE и связанных с ней инструментов в 2016 году.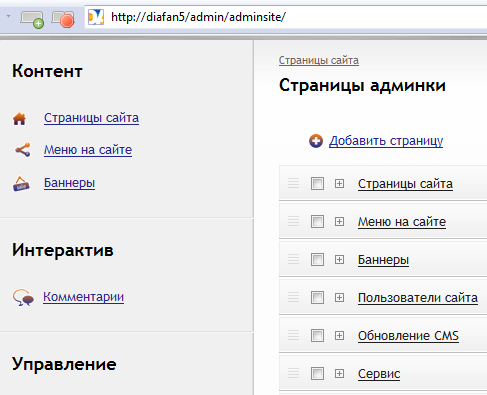 Нуклеиновые Кислоты Res. 44, D447–D456 [бесплатная статья PMC] [PubMed] [Google Scholar]
Нуклеиновые Кислоты Res. 44, D447–D456 [бесплатная статья PMC] [PubMed] [Google Scholar]
39. Перкинс Д. Н., Паппин Д. Дж., Кризи Д. М. и Коттрелл Дж. С. (1999) Вероятностная идентификация белков путем поиска в базах данных последовательностей с использованием данных масс-спектрометрии. Электрофорез 20, 3551–3567 [PubMed] [Google Scholar]
40. Келл Л., Кентербери Дж. Д., Уэстон Дж., Ноубл В. С. и МакКосс М. Дж. (2007) Полуконтролируемое обучение для идентификации пептидов из наборов данных протеомики дробовика. Нац. Методы 4, 923–925 [PubMed] [Google Scholar]
41. Бертольд М.Р., Цеброн Н., Дилл Ф., Габриэль Т.Р., Кёттер Т., Майнл Т., Ол П., Зиб К., Тиль К. и Висведель Б. (2008) KNIME: The Konstanz Information Miner. стр. 319–326, Springer Berlin Heidelberg, Berlin, Heidelberg [Google Scholar]
42.
Uszkoreit J., Maerkens A., Perez-Riverol Y., Meyer H.E., Marcus K., Stephan C., Kohlbacher O. и Eisenacher M. (2015) PIA: интуитивный механизм вывода белков с пользовательским веб-интерфейсом . Дж. Протеом Рез. 14, 2988–2997 [PubMed] [Google Scholar]
Дж. Протеом Рез. 14, 2988–2997 [PubMed] [Google Scholar]
43. Uszkoreit J., Perez-Riverol Y., Eggers B., Marcus K. и Eisenacher M. (2019) Вывод белков с использованием рабочих процессов PIA и стандартных форматов файлов PSI. Дж. Протеом Рез. 18, 741–747 [PubMed] [Google Scholar]
44. Чемберс М. К., Маклин Б., Берк Р., Амодей Д., Рудерман Д. Л., Нойманн С., Гатто Л., Фишер Б., Пратт Б., Эгертсон Дж., Хофф К., Кесснер Д., Тасман Н., Шульман Н., Фревен Б., Бейкер Т. А., Брусниак М. Ю., Паулс К., Кризи Д., Флешнер Л., Кани К., Молдинг К., Сеймур С. Л., Нувайсир Л. М., Лефевр Б., Кульманн Ф., Рорк Дж. ., Райнер П., Детлев С., Хеменуэй Т., Хамер А., Лэнгридж Дж., Коннолли Б., Чадик Т., Холли К., Экелс Дж., Дойч Э. В., Мориц Р. Л., Кац Дж. Э., Агус Д. Б., МакКосс М., Табб Д.Л. и Маллик П. (2012) Кроссплатформенный инструментарий для масс-спектрометрии и протеомики. Нац. Биотехнолог. 30, 918–920 [бесплатная статья PMC] [PubMed] [Google Scholar]
45.
Ким С., Мишериков Н. , Бандейра Н., Наварро Дж. Д., Вич Л., Мохаммед С., Хек А. Дж. и Певзнер П. А. (2010) Производящая функция пар CID, ETD и CID/ETD тандемных масс-спектров: приложения для поиска в базе данных. Мол. Клетка. протеомика
9, 2840–2852 [бесплатная статья PMC] [PubMed] [Google Scholar]
, Бандейра Н., Наварро Дж. Д., Вич Л., Мохаммед С., Хек А. Дж. и Певзнер П. А. (2010) Производящая функция пар CID, ETD и CID/ETD тандемных масс-спектров: приложения для поиска в базе данных. Мол. Клетка. протеомика
9, 2840–2852 [бесплатная статья PMC] [PubMed] [Google Scholar]
46. Крейг Р. и Бивис Р. К. (2004) ТАНДЕМ: сопоставление белков с тандемными масс-спектрами. Биоинформатика 20, 1466–1467 [PubMed] [Google Scholar]
47. Штейнберг Д., Дойч Э.В., Лам Х., Энг Дж.К., Сан З., Тасман Н., Мендоза Л., Мориц Р.Л., Эберсолд Р. и Несвижский А.И. (2011) iProphet: многоуровневый интегративный анализ протеомных данных дробовика улучшает скорость идентификации пептидов и белков и оценки ошибок. Мол. Клетка. протеомика 10, M111.007690 [бесплатная статья PMC] [PubMed] [Google Scholar]
48.
Рейтер Л., Клаассен М., Шримпф С.П., Йованович М., Шмидт А., Бухманн Дж.М., Хенгартнер М.О. и Эберсолд Р. (2009 г.) Частота ложных открытий при идентификации белков для очень больших наборов протеомных данных, полученных с помощью тандемной масс-спектрометрии. Мол. Клетка. протеомика
8, 2405–2417 [бесплатная статья PMC] [PubMed] [Google Scholar]
Мол. Клетка. протеомика
8, 2405–2417 [бесплатная статья PMC] [PubMed] [Google Scholar]
49. Лам Х., Дойч Э.В., Эддес Дж.С., Энг Дж.К., Штейн С.Е. и Эберсолд Р. (2008)Создание консенсусных спектральных библиотек для идентификации пептидов в протеомике. Нац. Методы 5, 873–875 [бесплатная статья PMC] [PubMed] [Google Scholar]
50. Телеман Дж., Рёст Х.Л., Розенбергер Г., Шмитт У., Мальмстрем Л., Мальмстрем Дж. и Левандер Ф. (2015) DIANA – алгоритмические усовершенствования для анализа независимых от данных данных сбора данных МС. Биоинформатика 31, 555–562 [PubMed] [Google Scholar]
51. Рёст Х.Л., Лю Ю., Д’Агостино Г., Занелла М., Наварро П., Розенбергер Г., Коллинз Б.К., Джиллет Л., Теста Г., Мальмстрём Л. и Эберсольд Р. (2016) TRIC: стратегия автоматического выравнивания для воспроизводимого количественного определения белка в целевой протеомике. Нац. Методы 13, 777–783 [бесплатная статья PMC] [PubMed] [Google Scholar]
52.
Вишневский Дж. Р., Хайн М. Ю., Кокс Дж. и Манн М. (2014) «Протеомная линейка» для оценки количества копий белка и концентрации без дополнительных стандартов. Мол. Клетка. протеомика
13, 3497–3506 [бесплатная статья PMC] [PubMed] [Google Scholar]
и Манн М. (2014) «Протеомная линейка» для оценки количества копий белка и концентрации без дополнительных стандартов. Мол. Клетка. протеомика
13, 3497–3506 [бесплатная статья PMC] [PubMed] [Google Scholar]
53. Брудерер Р., Бернхардт О.М., Ганди Т., Суан Ю., Зондерманн Дж., Шмидт М., Гомес-Варела Д. и Рейтер Л. (2017) Оптимизация экспериментальных параметров в независимой от данных масс-спектрометрии значительно увеличивает глубину и воспроизводимость результатов. Мол. Клетка. протеомика 16, 2296–2309 [бесплатная статья PMC] [PubMed] [Google Scholar]
54.
Tabb D.L., Vega-Montoto L., Rudnick P.A., Variyath A.M., Ham A.J., Bunk D.M., Kilpatrick L.E., Billheimer D. D., Blackman R.K., Cardasis H. L., Carr S.A., Clauser K.R., Jaffe J., Kowalski K.A., Neubert T., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., Regnier F., REGNE , Schilling B., Tegeler T.J., Wang M., Wang P., Whiteaker J.R., Zimmerman L.J.



 Там же можно добавить, удалить или дезактивировать номера телефонов). ВАЖНО: все номера телефонов должны быть в международном формате вида +79001234567.
Там же можно добавить, удалить или дезактивировать номера телефонов). ВАЖНО: все номера телефонов должны быть в международном формате вида +79001234567.