способы создать модель. Часть 1 / Хабр
Пожалуй, каждый или почти каждый читатель играл в современные графонистые игры, смотрел мультики Пиксар или хотя бы кино от Марвел или ДС. Или любой другой крупной компании — сейчас сложно найти фильмы без графики. И за просмотром или игрой наверняка задавались вопросом — а как это сделано? А, может, даже фантазировали, а что бы Вы сделали, если бы вдруг освоили 3D-графику?
Автор сообщества Фанерозой, биотехнолог, Людмила Хигерович.
Сегодня эта графика кажется уж слишком дешевой и корявой. Однако в год выхода фильма немногие киноленты могли похвастаться даже этим (опустим историю с хоббитами — она скорее исключение, чем правило). Кадр из фильма «Газонокосильщик», 1992, режиссер Бретт Леонард.
Что ж, ещё лет 30 — 40 назад графоний могли себе позволить только очень богатые компании с очень мощными суперкомпьютерами, а на создание одного ассета (набора функциональных моделей) уходили годы.
Осторожно! От этой малополигональной зарубы у некоторых читателей может случиться острый приступ ностальгии! «Готика» от Piranha bytes, 2001 год.
Сегодня не проходит и месяца, чтобы не была анонсирована какая-нибудь крутая (или не очень) игрушка с трассировкой лучей или киношка с полностью перерисованными фонами и героями. Скрины приводить не буду, т.к. Fair use работает через раз, а примеры вы и сами можете привести, даже побольше, чем я. Лучше скину картинку со своей любимой игрой из моего детства.
От полета в этой игре в облике дракона в начале двухтысячных захватывало дух! Сейчас графика сильно устарела, однако эффекты просвечивания все еще вызывают уважение. «Глаз Дракона» от издательства Акелла, 2002 год.
Софт для 3D-моделирования тоже не стоит на месте и развивается не только в сторону улучшения визуализации, но и в сторону friendly-функций — начиная с упрощения интерфейсов и функций и заканчивая оптимизацией производительности “под картошку”, что позволяет буквально каждому пользователю ПК подобрать себе софт по нуждам и возможностям. При желании можно и на телефон найти программу. Правда, функционал и качество большинства таких приложений оставляют желать лучшего.
Но при обилии программ в них легко запутаться. Не умаляет проблемы и избыток туториалов, хотя бы потому, что избыток этот иллюзорный.
- Во-первых, туториалов на одни программы явно больше, чем на другие. Обучающих видео по 3ds max или Zbrush много разного качества и толка, причём сейчас их несложно найти практически на любом языке. Тогда как на менее популярные программы есть десяток туториалов на английском, да и те от разработчиков оригинального софта без особых пояснений.
- Во-вторых, существует огромная пропасть между туториалами для “чайников” и “тех, кто что-то умеет или прошёл наш базовый курс/школу/читал/смотрел/пробовал” и так далее.
 При этом начинающие свой путь новички зачастую не любят открывать видео “для нубов”, но и во второй категории мало что могут понять, заваливая комментарии вопросами, а после ещё и обижаясь на автора туториала.
При этом начинающие свой путь новички зачастую не любят открывать видео “для нубов”, но и во второй категории мало что могут понять, заваливая комментарии вопросами, а после ещё и обижаясь на автора туториала. - В-третьих, почти все туториалы ставят конкретную цель. Да, это хорошо, когда в туториале есть определённый пример и можно сравнить свой результат с результатом автора. Но в большинстве своём это повод для разочарования. Я ни в коем случае не против этого, даже наоборот, сама пропагандирую. Однако как быть с теми, кто даже не знает, с чего начать? Да и стоит ли?
Что ж, хочу сказать, что многие программы заточены под определённый тип или подход к моделированию. Конечно, существуют пакеты или даже самостоятельные программы “10 в одном”, как тот же 3Ds max или Blender. Однако узкоспециализированных программ достаточно много.
Отдельный повод для самобичевания — работы, подписанные как “Вот первое, что я сделал.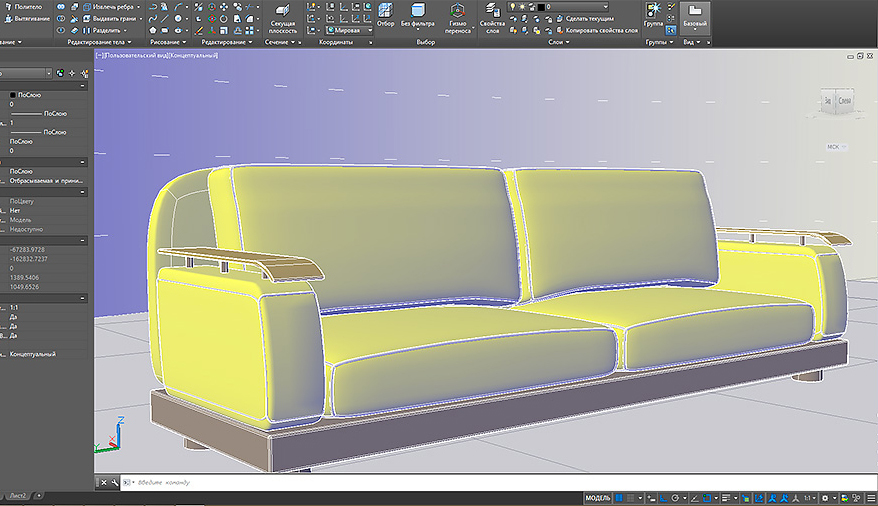
Этот дракон — одна из моих первых работ! — подписал автор статьи, выкладывая рендер на одном из сайтов в далеком 2012. Да, это первая работа в конкретно том софте и конкретно теми инструментами, но до нее было знакомство с простейшими объектами в 3ds max, лепка в корявом пробном режиме MQO, ковыряние в разрезанных и сшитых примитивах и кучка брошенных на полпути недоделок в виде голов, хвостов, лиц и прочего)
Поэтому я решила познакомить Вас не с основами моделирования как таковыми, и не с программами для моделирования, хотя упоминать конкретные примеры буду, это неизбежно.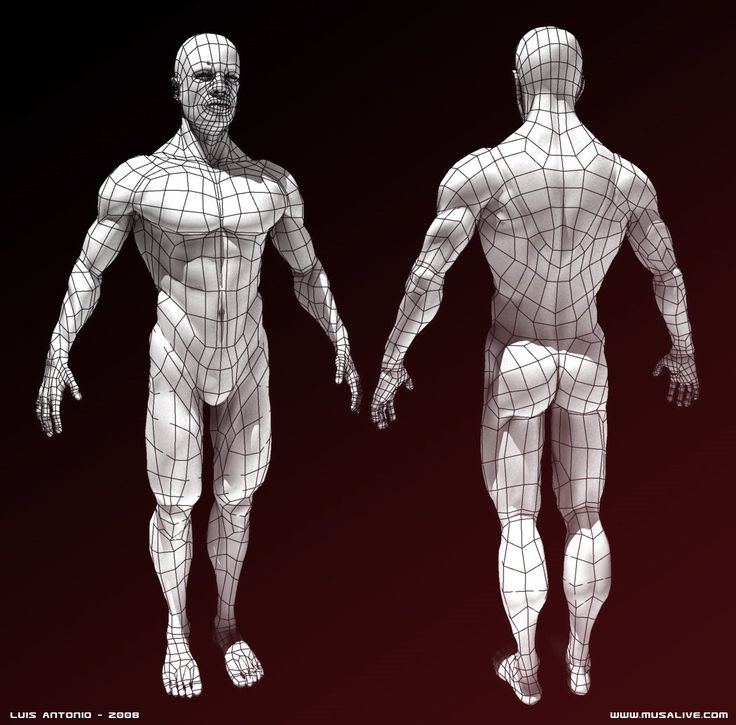 Я познакомлю вас с подходами к моделированию и методами создания моделей вне зависимости от конечного результата. Да, это всё было просто огромное лирическое отступление. Но сейчас перейдём к делу.
Я познакомлю вас с подходами к моделированию и методами создания моделей вне зависимости от конечного результата. Да, это всё было просто огромное лирическое отступление. Но сейчас перейдём к делу.
!Предупреждение!
▍ Подходы и методы
Существует два основных подхода к моделированию — объектный, также называемый векторным, и полигональный. Векторная 3D-графика строится на фиксированных формах (поверхностях геометрических объектов), представляющих собой совокупность множества точек поверхности или только информации о габаритах объекта (длина-ширина-высота, диаметр, объём, точки пересечения и т.п.). В некоторых случаях такой подход выгоден, например, в промышленном моделировании. Но порой работать в таком ключе становится сложно. Наложить на такой объект текстуры, например, не представляется возможным — вместо этого используют шейдеры (

Полигональное моделирование в самом просто его виде — создание полигонов через вершины:
Полигональное моделирование отличается тем, что поверхность объекта разбивается на точки — вершины (axis), соединяющие их “рёбра” и заполнение между ними — полигоны (polygons). Друг от друга полигоны ограничивают грани или рёбра (lines, ribs), соединяющие 2 вершины.
Совокупность вершин и полигонов называется мешем (
Полигоны можно разбивать (divide, subdivide), увеличивая детализацию и сглаживая грубые грани, можно сокращать (decimate) для экономии памяти компьютера, уменьшения нагрузки или упрощения работы.
В рамках этого подхода, модель в базовом своём виде состоит из меша и представляет собой объект или комплекс пересекающихся, или самостоятельных объектов, объединенных смыслом, функцией или единым финальным обликом.
Некоторые программы успешно совмещают векторное и полигональное моделирование или могут конвертировать (преобразовывать) один вид модели в другой, превращая облако точек поверхности в вершины или наоборот. К таким программам, например, относятся Autocad и Blender.
Кроме вышеназванных, существуют другие подходы, вроде математического программирования. Но на них мы останавливаться не будем — они слишком узкоспециализированы и используются в основном в визуализации формул и графиков.
Сразу скажем, что сосредоточим внимание на полигональном моделировании, так как оно более распространено, для работы с ним больше софта и оно куда более востребовано — полигональные модели используются в играх, мультфильмах, фильмах, для печати фигурок, артов и прочего. При этом моделировать даже в рамках полигонального подхода можно по-разному, причём получая очень близкий по виду конечный результат.
▍ Моделируем, как можем
Итак, начнём с самого простого по навыку, но одного из сложных технически — 3D-сканирование. Фактически, от человека тут мало что зависит, некоторые профессионалы даже не считают этот способ настоящим моделированием.
Фактически, от человека тут мало что зависит, некоторые профессионалы даже не считают этот способ настоящим моделированием.
Суть его в том, что создаётся множество фотографий объекта или помещения на одном и том же расстоянии, но под разными углами. После этого специальная программа анализирует фотографии и создаёт облако точек, а затем — меши. Многие из них ещё и накладывают текстуру, сформированную из фотографий.
Правда, чтобы получить хорошее качество, нужна хорошая камера, желательно лазерный сканер, мощный компьютер и специальная программа. Впрочем, есть и бюджетные версии — некоторые программы для фотограмметрии вполне совместимы с современным телефоном, и могут быть использованы на домашнем компьютере. Но будьте готовы делать по 600 фотографий одной и той же детали с разных сторон и ждать 20 часов, пока ваша модель скомпилируется. И не забудьте про расстояние — все точки фотографирования должны быть равноудалены от объекта, иначе последний будет перекошен. Ну и для работы потребуется “допилить” модель — “починить” дыры в меше, отрезать куски ненужного фона, поправить UV и т. д.
д.
Отдельный метод на границе сканирования и объектного моделирования мы рассмотрим позднее. Скажем только, что с его помощью актёров из реальной жизни переносят в фильмы и игры.
Второй способ — “рисование” полигонов. Вы буквально берёте и рисуете грани, вершины и полигоны, подобно черчению, сбору мозаики или аппликации. Таким образом можно получить очень точный результат, особенно когда требуется сделать малополигональную модельку точно по концепту. В этом случае в некоторых программах можно разместить картинку с примером и “чертить” буквально на ней. Однако полную и подробную модельку таким способом не сделаешь. Другое дело — создание болванок для последующей лепки.
- Плюсы: точность, простота.
- Минусы: долго, мало полигонов.
Так выглядит немного урезанный процесс создания модели по полигонам. Это может пригодиться, например, когда есть четкий векторный рисунок или силуэт, который не требует большой детализации
Третий способ — примитивы. Собственно, обычно с него все и начинают, так как набор базовых фигур (primitives) есть в каждой программе. На рабочем поле размещают примитив или несколько примитивов, составляют из них композицию, деформируют, режут и сливают. Здесь же можно производить булевые операции (boolean operation). Возможно, вы уже слышали про это в рамках математики. Если нет, то выглядит это следующим образом: мы можем складывать и вычитать геометрию из одного объекта другим. Так, цилиндрами мы можем наделать отверстий в кубе, или сделать квадратное окно в сфере.
Собственно, обычно с него все и начинают, так как набор базовых фигур (primitives) есть в каждой программе. На рабочем поле размещают примитив или несколько примитивов, составляют из них композицию, деформируют, режут и сливают. Здесь же можно производить булевые операции (boolean operation). Возможно, вы уже слышали про это в рамках математики. Если нет, то выглядит это следующим образом: мы можем складывать и вычитать геометрию из одного объекта другим. Так, цилиндрами мы можем наделать отверстий в кубе, или сделать квадратное окно в сфере.
- Плюсы: простота работы, булевые операции.
- Минусы: низкая точность, грубые формы.
Объектное моделирование — идеальный вариант для создания антенн, машин, механизмом — словом, для разнообразных твердотельных и технических моделей (т.н. Hard surface). Впрочем, для органики, порой тоже приходится использовать нечто подобное — например, для создания глаз.
Когда-то на заре моделирования, этот и предыдущий — были единственными способами полигонального моделирования, и отнимали кучу времени для приведения в порядок.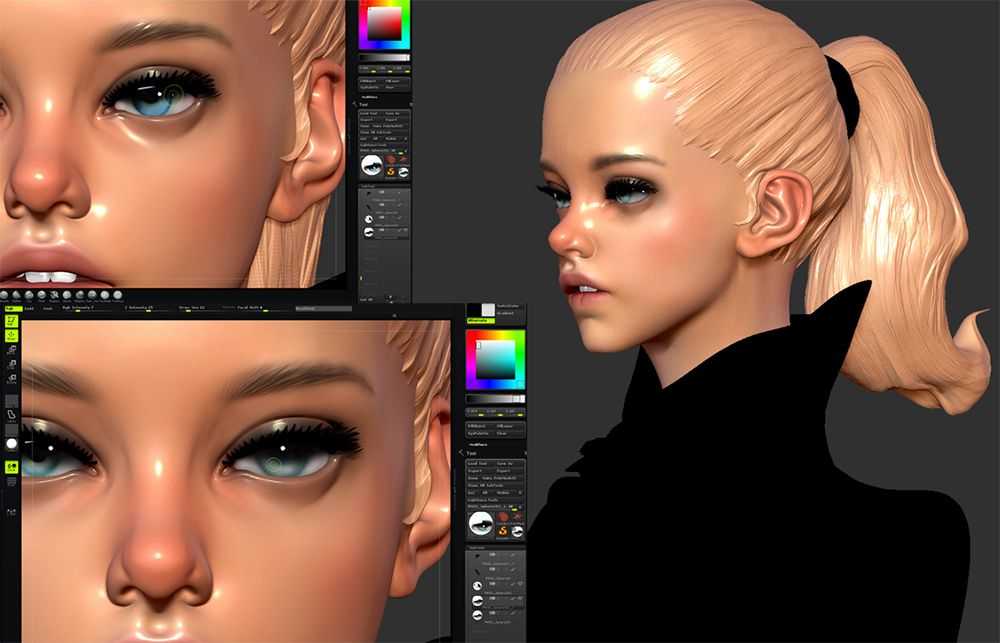 Но время шло, появлялись новые способы и средства визуализации.
Но время шло, появлялись новые способы и средства визуализации.
Скульптурирование или лепка (sculpting). Откровенно говоря, самый любимый метод моделирования объектов у автора статьи. Суть его заключается в том, что из базовой формы (примитива) по принципу куска глины или пластилина лепится новая форма с помощью выдавливания и наращивания объёма. Крупные куски отсекаются, тонкими инструментами создаётся мелкая детализация — совсем как в реальном скульптурировании.
Быстрый скульпт базовой морды в Zbrush. На данный момент зебра — самый мощный и функциональный софт для скульптинга. Разумеется, аналоги есть, и они добавлены в каждую крупную программу для моделирования, однако полностью заменить и вытеснить зебру они пока не могут.
Есть также и пограничные методы, использующие сразу несколько технологий, например, скелетное моделирование или альфа-проекция. Однако это может оказаться весьма сложным для понимания, и возможно заслуживает более подробного анализа и представления, чем мы можем позволить себе здесь.
Скелетное моделирование присутствует в разных программах, и реализовано в каждой по-своему. Так, например, выглядит скелетное моделирование в ZBrush — мы создаем какие-то базовые формы (основу, скелет) из Зсфер (Zsphere), и обращиваем ее «кожей». «Мясистость» контролируется величиной костей и соединяющих их переходов. После создания кожи ее так же можно модифицировать, подобно тому, как модифицировали бы любой полигональный объект.
Всё это очень интересно, но как же определиться с применением? Что если я хочу, скажем, освоить только определённую технологию для вполне конкретной цели? Например, создавать исключительно персонажей для игр? Или наоборот, переносить на большие экраны свои или чужие фантазии? А, может, я прирождённый техник, и мечтаю печатать на 3D-принтере свои механизмы?
Что ж, придётся показать, на что способно 3D-моделирование на практике, заодно демонстрируя конкретные примеры и методы, так сказать, наглядно.
Однако это придётся отложить на следующий длиннопост, так как примеров масса, а этот текст, итак, раздулся. Поэтому, до новых Встреч!
Поэтому, до новых Встреч!
Все картинки в посте, кроме скриншотов игр и кадра из фильма — авторские. Модели также созданы автором текста.
Создание 3D моделей обзор, сравнение, лучшие продукты, внедрения, поставщики.
3D-моделирование — процесс создания трёхмерной модели объекта. Задача 3D-моделирования — разработать зрительный объёмный образ желаемого объекта. При этом модель может как соответствовать объектам из реального мира (автомобили, здания, ураган, астероид), так и быть полностью абстрактной (проекция четырёхмерного фрактала).
Графическое изображение трёхмерных объектов отличается тем, что включает построение геометрической проекции трёхмерной модели сцены на плоскость (например, экран компьютера) с помощью специализированных программ. Однако с созданием и внедрением 3D-дисплеев и 3D-принтеров трёхмерная графика не обязательно включает в себя проецирование на плоскость.
В трехмерной компьютерной графике создание 3D моделей — это процесс разработки математического представления любой поверхности объекта (неодушевленного или живого) в трех измерениях с помощью специализированного программного обеспечения.
3D-модели широко используются везде в 3D-графике и САПР. Этапы создания 3D-модели:
- моделирование — создание трёхмерной математической модели сцены и объектов в ней;
- текстурирование — назначение поверхностям моделей растровых или процедурных текстур;
- освещение — установка и настройка источников света;
- анимация (в некоторых случаях) — придание движения объектам;
- динамическая симуляция (в некоторых случаях) — автоматический расчёт взаимодействия частиц, твёрдых/мягких тел и пр. с моделируемыми силами гравитации, ветра, выталкивания и др. и друг с другом;
- рендеринг (визуализация) — построение проекции в соответствии с выбранной физической моделью;
- композитинг (компоновка) — доработка изображения;
- вывод полученного изображения на устройство вывода — дисплей или 3D-принтер.
Создание трехмерных моделей применяется в:
- В разработке компьютерных игр;
- Как один из этапов 3D-визуализации;
- Для предварительной оценки технических свойств проектируемого изделия;
- В подготовке образцов к 3D-печати;
- Для создания прототипов изделий.

В последнее время создание трехмерной модели является обязательным этапом производственного процесса, поскольку позволяет в деталях оценить проектируемые образцы. В дальнейшем эта модель пригодится для изготовления прототипов и макетов создаваемых изделий.
- Продукты
- Сравнение
- Поставщики
- Производители
- FAQ
- Материалы
Поставщики Создание 3D моделей
Производители Создание 3D моделей
F.
 A.Q.
Создание 3D моделей
A.Q.
Создание 3D моделей- Проектирование моделей в результате дает не просто картинку, но и готовый шаблон для изготовления при помощи 3d печати или фрезерования.
- Разработка 3d моделей подразумевает высокую точность. Если речь идет о мелких деталях, которые впоследствии должны взаимодействовать между собой, создание трехмерных прототипов с использованием специальных программ позволят просчитать размеры вплоть до микронов.
- Изготовление 3d моделей даже без последующего воплощения найдут применение. Например, полезны такие 3d модели для дизайнеров, благодаря возможности «повертеть» какой-нибудь стол в каталоге, получить больше информации об объекте и повлиять на решение о покупке.
Создание чертежа изделия по-прежнему используется в промышленности. Однако большинство индустрий переходит к тому, что выбирает создание 3d объектов как точный и надежный вариант.
В 3D-печати используется четыре основных типа программного обеспечения, которые выполняют свои функции последовательно.
Моделирование. Программное обеспечение для 3D-моделирования позволяет создавать 3D-модели на компьютере. Вы можете спроектировать что угодно, от отвертки до модели автомобиля.
Конечно, если вы не хотите создавать собственную 3D-модель, вы можете скачать 3D-модели сделанные другими.
Редактирование. Просмотр, редактирование и ремонт моделей для печати в формате STL. Независимо от того, создаете ли вы свою собственную 3D-модель или загружаете ее из интернета, чаще всего вы получаете STL-файл. Однако не все файлы STL идеальны, а некоторые могут иметь и явные недостатки. Дефекты моделей приводят к браку или сбоям при печати. Программное обеспечение просмотра, редактирования и ремонта файлов STL позволяет визуализировать, изменять и исправлять файлы STL, чтобы гарантировать, что они готовы к 3D-печати. Обычно такое ПО совмещено со следующей разновидностью программ для 3D-печати — слайсерами.
Нарезка на слои. Слайсер преобразует STL-файл в G-код — управляющий код для принтера, содержащий команды для печати каждого слоя модели и последовательность их применения. Зачастую слайсер содержит в себе функции диагностики и ремонта или автоисправления моделей.
Зачастую слайсер содержит в себе функции диагностики и ремонта или автоисправления моделей.
Калибровка. Программа для калибровки помогает откалибровать принтер и упрощает подбор параметров для оптимальной печати. Она нужна как при первоначальной настройке принтера, так и в процессе использования — для определения параметров печати конкретного пластика.
Печать. Хост — программа для печати, она отвечает за передачу G-кода на принтер, позволяет отслеживать процесс печати и изменять настройки в режиме реального времени, если принтер подключен к компьютеру. Многие принтеры имеют встроенное ПО, печатающее модели с флешки или карты памяти.
Часто функционал слайсера и хоста объединен в одной программе.
Благодаря этими четырем типами программного обеспечения создание модели для 3D печати воплощается в физический 3D-объект.
Вы уверены что хотите удалить ?
Да
Нет
Создание модели данных в Excel
Модель данных позволяет интегрировать данные из нескольких таблиц, эффективно создавая реляционный источник данных в книге Excel. В Excel модели данных используются прозрачно, предоставляя табличные данные, используемые в сводных таблицах и сводных диаграммах. Модель данных визуализируются как коллекция таблиц в списке полей, и в большинстве раз вы даже не узнаете, что она существует.
В Excel модели данных используются прозрачно, предоставляя табличные данные, используемые в сводных таблицах и сводных диаграммах. Модель данных визуализируются как коллекция таблиц в списке полей, и в большинстве раз вы даже не узнаете, что она существует.
Прежде чем приступить к работе с моделью данных, необходимо получить некоторые данные. Для этого мы будем использовать интерфейс Get & Transform (Power Query), поэтому вам может потребоваться выполнить шаг назад и посмотреть видео, или следуйте нашему руководству по обучению по get & Transform и Power Pivot.
Где есть Power Pivot?
-
Excel 2016 & Excel для Microsoft 365 — Power Pivot включен в ленту.
org/ListItem»>
-
Excel 2010 — скачайте надстройку Power Pivot, а затем установите надстройку Power Pivot.
Excel 2013 — Power Pivot входит в Office профессиональный плюс Excel 2013, но не включен по умолчанию. Дополнительные сведения о запуске надстройки Power Pivot для Excel 2013.
Где находится get & Transform (Power Query)?
-
Excel 2016 & Excel для Microsoft 365 . Get & Transform (Power Query) интегрировано с Excel на вкладке «Данные«.
org/ListItem»>
-
Excel 2010 — скачивание и установка Power Query надстройки.. После активации на ленту Power Query вкладки.
Excel 2013 — Power Query — это надстройка, которая входит в Excel, но ее необходимо активировать. Перейдите к разделу «Параметры >» > надстроек, а затем в раскрывающемся списке «Управление» в нижней части панели выберите com-надстройки > Go. Проверьте microsoft Power Query Excel, а затем ОК, чтобы активировать его. На Power Query будет добавлена вкладка Power Query.
Начало работы
Сначала необходимо получить некоторые данные.
В Excel 2016 и Excel для Microsoft 365 используйте data >Get & Transform Data > Get Data > Get Data to import data from any number of external data sources, such as a text file, Excel workbook, website, Microsoft Access, SQL Server, or another relational database that contains multiple related tables.
В Excel 2013 и 2010 перейдите к Power Query >получения внешних данных и выберите источник данных.
Excel предложит выбрать таблицу. Если вы хотите получить несколько таблиц из одного источника данных, установите флажок «Включить выбор нескольких таблиц «. При выборе нескольких таблиц Excel автоматически создает модель данных.
Примечание: В этих примерах мы используем книгу Excel с вымышленными сведениями о классах и оценках учащихся. Вы можете скачать пример книги модели данных учащихся и следовать инструкциям. Вы также можете скачать версию с готовой моделью данных..
Вы можете скачать пример книги модели данных учащихся и следовать инструкциям. Вы также можете скачать версию с готовой моделью данных..
Выберите одну или несколько таблиц и нажмите кнопку «Загрузить «.
Если необходимо изменить исходные данные, можно выбрать параметр «Изменить «. Дополнительные сведения см. в статье «Общие сведения Редактор запросов (Power Query)».
Теперь у вас есть модель данных, которая содержит все импортированные таблицы, и они будут отображаться в списке полей сводной таблицы.
Примечания:
-
Модели создаются неявно, когда вы импортируете в Excel несколько таблиц одновременно.
org/ListItem»>
-
Модель может содержать одну таблицу. Чтобы создать модель на основе только одной таблицы, выберите таблицу и нажмите кнопку Добавить в модель данных в Power Pivot. Это может понадобиться в том случае, если вы хотите использовать функции Power Pivot, например отфильтрованные наборы данных, вычисляемые столбцы, вычисляемые поля, ключевые показатели эффективности и иерархии.
org/ListItem»>
-
Советы по сокращению размера модели данных см. в статье «Создание модели данных, оптимизированной для памяти, с помощью Excel и Power Pivot».
-
Дополнительные сведения см. в руководстве по импорту данных в Excel и созданию модели данных.
Модели создаются явно, если вы импортируете данные с помощью надстройки Power Pivot. В надстройке модель представлена в макете с вкладками, аналогичном Excel, где каждая вкладка содержит табличные данные. Дополнительные сведения об импорте данных с помощью надстройки Power Pivotсм. в статье «Получение данных с помощью SQL Server данных».
Связи между таблицами могут создаваться автоматически при импорте связанных таблиц, у которых есть связи по первичному и внешнему ключу. Excel обычно может использовать импортированные данные о связях в качестве основы для связей между таблицами в модели данных.
Совет: Как определить, есть ли в книге модель данных? Перейдите Power Pivot > Управление. Если вы видите данные, как на листе, то модель существует. См. дополнительные сведения о том, какие источники данных используются в модели данных книги.
Если вы видите данные, как на листе, то модель существует. См. дополнительные сведения о том, какие источники данных используются в модели данных книги.
Создание связей между таблицами
Следующим шагом является создание связей между таблицами, чтобы вы могли извлекать данные из любой из них. Каждая таблица должна иметь первичный ключ или уникальный идентификатор поля, например идентификатор учащегося или номер класса. Самый простой способ — перетащить эти поля, чтобы подключить их в представлении схемы Power Pivot.
-
Перейдите в power Pivot > Manage.
-
На вкладке « Главная» выберите » Представление схемы».

-
Будут отображены все импортированные таблицы, и может потребоваться некоторое время, чтобы изменить их размер в зависимости от количества полей в каждой из них.
-
Затем перетащите поле первичного ключа из одной таблицы в следующую. В следующем примере показано представление схемы таблиц учащихся.
Мы создали следующие ссылки:
-
tbl_Students | Идентификатор учащегося > tbl_Grades | Идентификатор учащегося
Другими словами, перетащите поле «Идентификатор учащегося» из таблицы «Учащиеся» в поле «Идентификатор учащегося» в таблице «Оценки».

-
tbl_Semesters | Идентификаторы > tbl_Grades | Семестр
-
tbl_Classes | Номер класса > tbl_Grades | Номер класса
Примечания:
-
Имена полей не обязательно должны совпадать для создания связи, но они должны быть одинаковыми типами данных.
-
Соединители в представлении схемы имеют «1» с одной стороны, а «*» — с другой. Это означает, что между таблицами существует связь «один ко многим», которая определяет, как данные используются в сводных таблицах. См. дополнительные сведения о связях между таблицами в модели данных.
-
Соединители указывают только на наличие связи между таблицами.
 На самом деле они не показывают, какие поля связаны друг с другом. Чтобы просмотреть ссылки, перейдите в раздел Power Pivot > Manage > Design > Relationships > Управление связями. В Excel можно перейти к разделу «>данных».
На самом деле они не показывают, какие поля связаны друг с другом. Чтобы просмотреть ссылки, перейдите в раздел Power Pivot > Manage > Design > Relationships > Управление связями. В Excel можно перейти к разделу «>данных».
-
Создание сводной таблицы или сводной диаграммы с помощью модели данных
Книга Excel может содержать только одну модель данных, но эта модель может содержать несколько таблиц, которые можно многократно использовать в книге. Вы можете добавить дополнительные таблицы в существующую модель данных в любое время.
-
В Power Pivotперейдите к разделу » Управление».
org/ListItem»>
-
Выберите место размещения сводной таблицы: новый лист или текущее расположение.
-
Нажмите кнопку «ОК», и Excel добавит пустую сводную таблицу с областью списка полей справа.
На вкладке « Главная» выберите сводную таблицу.
Затем создайте сводную таблицу или сводную диаграмму. Если вы уже создали связи между таблицами, можно использовать любое из их полей в сводной таблице. Мы уже создали связи в образце книги модели данных учащихся.
Добавление имеющихся несвязанных данных в модель данных
Предположим, вы импортировали или скопировали много данных, которые вы хотите использовать в модели, но не добавили их в модель данных. Принудительно отправить новые данные в модель очень просто.
Принудительно отправить новые данные в модель очень просто.
-
Начните с выбора любой ячейки в данных, которые необходимо добавить в модель. Это может быть любой диапазон данных, но лучше всего использовать данные, отформатированные в виде таблицы Excel .
-
Добавьте данные одним из следующих способов.
-
Щелкните Power Pivot > Добавить в модель данных.
-
Выберите Вставка > Сводная таблица и установите флажок Добавить эти данные в модель данных в диалоговом окне «Создание сводной таблицы».

Диапазон или таблица будут добавлены в модель как связанная таблица. Дополнительные сведения о работе со связанными таблицами в модели см. в статье Добавление данных с помощью связанных таблиц Excel в Power Pivot.
Добавление данных в таблицу Power Pivot данных
В Power Pivot невозможно добавить строку в таблицу, введя текст непосредственно в новой строке, как это можно сделать на листе Excel. Но можно добавить строки , скопируйте и вставьте или обновите исходные данные и обновите модель Power Pivot.
Дополнительные сведения
Вы всегда можете задать вопрос специалисту Excel Tech Community или попросить помощи в сообществе Answers community.
См. также
Ознакомьтесь & по преобразованию и обучению Power Pivot
Общие сведения о редакторе запросов (Power Query)
Создание модели данных, оптимизированной для памяти, с помощью Excel и Power Pivot
Руководство. Импорт данных в Excel и создание модели данных
Импорт данных в Excel и создание модели данных
Определение источников данных, используемых в модели данных книги
Связи между таблицами в модели данных
Создание модели прогнозирования — AI Builder
Обратная связь
Twitter LinkedIn Facebook Адрес электронной почты
- Статья
- Чтение занимает 3 мин
В этом примере создается модель ИИ прогноза Power Apps, использующая таблицу намерений интернет-покупателей в Microsoft Dataverse. Чтобы получить эти образцы данных в свою среду Microsoft Power Platform, включите параметр Развертывать примеры приложений и данных при создании среды, как описано в разделе Сборка модели в AI Builder. Или выполните более подробные инструкции в разделе Подготовка данных. После добавления образца данных в Dataverse выполните следующие действия, чтобы создать модель.
Чтобы получить эти образцы данных в свою среду Microsoft Power Platform, включите параметр Развертывать примеры приложений и данных при создании среды, как описано в разделе Сборка модели в AI Builder. Или выполните более подробные инструкции в разделе Подготовка данных. После добавления образца данных в Dataverse выполните следующие действия, чтобы создать модель.
Войдите в Power Apps, а затем выберите AI Builder > Исследовать.
Выберите Прогнозирование. Введите имя для этой модели, а затем выберите команду Создать.
Выбор исторических результатов
Представьте себе прогноз, который требуется создать в AI Builder. Например, в случае вопроса «Будет ли клиент уходить?» подумайте о таких факторах:
- Где находится таблица, содержащая сведения об оттоке клиентов?
- Есть ли столбец, в котором конкретно указано, ушел ли клиент?
- Имеются ли в столбце неизвестные, возможно создающие неопределенность?
Используйте эти сведения для выбора. При работе с предоставленными примерами данных вопрос будет таким: «Пользователь моего интернет-магазина сделал покупку?» Если сделал, то для этого клиента должен быть зарегистрирован доход. Таким образом, наличие дохода от этого клиента и будет историческим результатом. Если эта информация пуста, то AI Builder может помочь вам сделать прогноз.
При работе с предоставленными примерами данных вопрос будет таким: «Пользователь моего интернет-магазина сделал покупку?» Если сделал, то для этого клиента должен быть зарегистрирован доход. Таким образом, наличие дохода от этого клиента и будет историческим результатом. Если эта информация пуста, то AI Builder может помочь вам сделать прогноз.
В раскрывающемся меню Таблица выберите таблицу, содержащую данные, и результат, который требуется спрогнозировать. В образце данных выберите Намерение интернет-покупателя.
В раскрывающемся меню Столбец выберите столбец, содержащий результат. В образце данных выберите Доход (метка). Если вы хотите попробовать предсказать число, выберите ExitRates.
Если вы выбрали набор параметров, который содержит несколько результатов, возможно, потребуется соотнести его с ответом «Да» или «Нет», чтобы предсказать, произойдет нечто или нет.

Если вы хотите спрогнозировать несколько результатов, используйте в примере бразильский набор данных электронной торговли и выберите Заказ BC в раскрывающемся меню Таблица и Сроки доставки в раскрывающемся меню Столбец.
Примечание
AI Builder поддерживает следующие типы данных для столбца результатов:
- Да/нет
- Варианты
- Целое число
- Десятичное число
- Число с плавающей запятой
- Валюта
Выбор столбцов данных для обучения модели
После выбора Таблица и Столбец и сопоставления результата можно внести изменения в столбцы данных, используемые для обучения модели. По умолчанию выбраны все соответствующие столбцы. Можно отменить выбор столбцов, которые могут уменьшить точность модели. Если вы не уверены, что здесь делать, не волнуйтесь. AI Builder попытается найти столбцы, предоставляющие оптимальную модель. В примере данных просто оставьте все как есть и выберите Далее.
В примере данных просто оставьте все как есть и выберите Далее.
Рекомендации по выбору столбцов данных
Учтите самое важное: не определяется ли результатом какой-то столбец, который не является столбцом исторического результата.
Предположим, вы хотите спрогнозировать, будет ли задерживаться доставка. В ваших данных может присутствовать фактическая дата доставки. Она появится только после доставки заказа. Если этот столбец включен, то модель будет иметь точность, близкую к 100 процентам. Заказы, которые требуют прогноза, пока еще не доставлены. Следовательно, столбец даты их доставки не будет заполнен. Поэтому стоит отменить выбор таких столбцов перед обучением. В машинном обучении это называется целевой утечкой или утечкой данных. AI Builder пытается отфильтровать столбцы, которые имеют слишком хорошее значение, однако обязательно проверьте их сами.
Примечание
При выборе полей данных некоторые типы данных, — например изображения, которые нельзя использовать в качестве входных для обучения модели — не отображаются. Кроме того, системные столбцы, такие как «Создано», исключаются по умолчанию.
Кроме того, системные столбцы, такие как «Создано», исключаются по умолчанию.
Если у вас есть связанные таблицы, которые могут повысить производительность прогноза, их можно также включить. Как и в случае, когда вы хотели предсказать, уйдет ли клиент, вам следует включить дополнительную информацию, которая может быть в отдельной таблице. В настоящее время AI Builder поддерживает связи «многие к одному».
Фильтрация данных
После выбора столбцов данных для обучения можно выполнить фильтрацию по данным. Ваши таблицы будут содержать все строки. Тем не менее может потребоваться сосредоточиться на обучении и прогнозировании подмножества строк. Если известно, что в одной таблице, используемой для обучения модели, есть нежелательные данные, можно использовать этот шаг, чтобы отфильтровать модель.
Например, если применить фильтр для просмотра только региона США, модель будет обучаться по строкам, в которых результат известен только для региона США. При обучении этой модели будет создаваться только прогноз для строк, в которых результат неизвестен только для региона США.
Фильтрация аналогична функции редактора представлений Power Apps. Начните с добавления:
- строка, содержащая одно условие фильтра;
- группа, которая позволяет вкладывать условия фильтра;
- Связанная таблица, которая позволяет создать условие фильтра для связанной таблицы.
Выберите столбец, оператор и значение, представляющее условие фильтра. Чтобы группировать строки или массово их удалять, можно использовать флажки.
Следующий шаг
Обучение и публикация модели прогнозирования
Примечание
Каковы ваши предпочтения в отношении языка документации? Пройдите краткий опрос (обратите внимание, что этот опрос представлен на английском языке).
Опрос займет около семи минут. Личные данные не собираются (заявление о конфиденциальности).
Обратная связь
Отправить и просмотреть отзыв по
Этот продукт Эта страница
Просмотреть все отзывы по странице
Создание модели геообработки—ArcGIS Pro | Документация
Модель – это визуальное представление рабочего процесса, где последовательно запускаются несколько инструментов геообработки. Выходные данные одного инструмента часто становятся входными для другого. В модели геообработки рабочий процесс представлен в виде диаграммы. Он также запускает описанный рабочий процесс. Вы можете использовать модель для многих целей, например:
Выходные данные одного инструмента часто становятся входными для другого. В модели геообработки рабочий процесс представлен в виде диаграммы. Он также запускает описанный рабочий процесс. Вы можете использовать модель для многих целей, например:
- Автоматизации повторяющихся задач
- Изучения различных выходных данных, полученных с применением различных наборов данных и параметров
- Визуального документирования методологии геообработки
- Постепенной разработки и совершенствования рабочих процессов
- Для распространения своего опыта и разработанных методов среди коллег
Обзор
Make a geoprocessing model overview video
- Продолжительность видео: 3:07.
- Это видео было создано с помощью версии ArcGIS Pro 2.3.
В этом руководстве вы создадите и запустите модель геообработки. Ваша модель автоматизирует рабочий процесс анализа из руководства Использование инструментов геообработки. В этом руководстве подразумевается, что вы уже знакомы с этим рабочим процессом.
В этом руководстве подразумевается, что вы уже знакомы с этим рабочим процессом.
- Предполагаемое время: 60 минут
- Требования к программному обеспечению: ArcGIS Pro
Открытие проекта
Пастбищам Новой Зеландии угрожают различные инвазивные виды. Эти злаковые распространяются преимущественно естественным способом, например, переносом семян по ветру, но также и через контакт с человеком. В руководстве Использование инструментов геообработки, вы анализировали близость одного из видов инвазивных трав (Nassella tussock) к потенциальному источнику контакта с человеком (площадки для кемпинга).
В этом руководстве вы создадите инструмент геообработки, который позволит вам применить такой же анализ для других инвазивных видов растений и иных антропогенных факторов.
- Запустите ArcGIS Pro и выполните вход, если необходимо.
- На начальной странице под разделом с недавними проектами щелкните Открыть другой проект.

- В диалоговом окне Открыть проект в разделе Портал нажмите ArcGIS Online .
- Вверху диалогового окна в поле Поиск введите Make a geoprocessing model tutorial и нажмите Enter.
- В списке результатов поиска щелкните Make a geoprocessing model, чтобы выбрать пакет проекта.
- Нажмите OK.
Изучение карты
Карта содержит несколько слоев с местоположениями, где человек может контактировать с инвазивными растениями: платные кемпинги, площадки для отдыха, железные и автодороги, населенные места. Карта также содержит слои четырех видов инвазивных трав: Nassella tussock, паслен окаймленный, темеда австралийская и чилийский ковыль.
Слои инвазивных видов отображает границы, в пределах которых было найдено данное растение. Они не показывают плотность растений в данном месте. Обозначенная территория может быть сильно заражена или на ней может встречаться только несколько растений.
- Щелкните вкладку Вид на ленте.
 В группе Окна щелкните Сбросить панели и выберите Сбросить панели для картографирования (по умолчанию).
В группе Окна щелкните Сбросить панели и выберите Сбросить панели для картографирования (по умолчанию).При этом открываются панели Содержание и Каталог, остальные панели остаются закрытыми.
- На панели Содержание включайте и выключайте слои различных инвазивных видов.
- На вкладке Карта щелкните Закладки . С помощью закладок вы более внимательно изучите каждый инвазивный вид.
- Включайте и выключайте слои с местами возможных контактов в комбинации со слоями различных инвазивных видов.
- По окончании изучения карты щелкните на вкладке Карта Закладки и выберите Marlborough Region.
- На панели Содержание включите следующие пять слоев и выключите все остальные:
- Basemap Labels
- Commercial Campgrounds
- Nassella Tussock Range
- Регион
- Базовая карта
Создание модели и добавление процесса
Вы создадите модель и добавите к ней процесс. Процесс – это инструмент геообработки, соединенный с входными и выходными данными. Модель с одним процессом является наиболее простой.
Процесс – это инструмент геообработки, соединенный с входными и выходными данными. Модель с одним процессом является наиболее простой.
- Щелкните вкладку Анализ на ленте. В группе Геообработка щелкните ModelBuilder .
В проекте откроется пустое окно модели. На ленте появится вкладка ModelBuilder.
- С панели Содержание перетащите слой Commercial Campgrounds в вид модели.
- В виде модели щелкните элемент Commercial Campgrounds правой кнопкой мыши и выберите Открыть.
- Нажмите OK в диалоговом окне Commercial Campgrounds.
- На ленте во вкладке ModelBuilder в группе Вставка щелкните Инструменты . (Щелкните значок, не стрелку)
Откроется панель Геообработка.
- На панели Геообработка, на вкладке Избранное, перетащите инструмент Буфер в вид модели рядом с Commercial Campgrounds.
- Задержите курсор мыши над входной переменной Commercial Campgrounds.
 Щелкните и перетащите соединительную линию к элементу инструмента Буфер. Отпустите кнопку мыши и нажмите Ввести объекты во всплывающем меню.
Щелкните и перетащите соединительную линию к элементу инструмента Буфер. Отпустите кнопку мыши и нажмите Ввести объекты во всплывающем меню. - Щёлкните правой кнопкой мыши инструмент Буфер и выберитеОткрыть.
- В диалоговом окне Буфер для параметра Расстояние [значение или поле] введите 5. Измените единицы измерения на Километры.
- Нажмите OK.
- Щелкните правой кнопкой на зеленой переменной выходных данных, CommercialCampgrounds_Buffer, и выбрите Добавить к карте.
Запустите модель из окна модели
В результате выполнения модели будут созданы 5-километровые буферы вокруг площадок для кемпинга. Это не является окончательной целью вашего исследования, но вы запустите модель сейчас, чтобы убедиться, что она работает как вы ожидаете. При запуске модули элемент инструмента Буфер станет красным на время обработки входных данных.
- На ленте во вкладке ModelBuilder в группе Запустить щелкните Запустить .

Когда модель закончит работу, появится сообщение об успешном выполнении.
- Закройте окно сообщения Модель.
- Сделайте активным вид карты Marlborough.
- На панели Содержание перетащите слой Commercial Campgrounds в верхнюю часть списка слоев.
- На панели Содержание щелкните правой кнопкой мыши слой ModelBuilder (который содержит буферный слой) и щелкните Удалить .
- На панели Каталог перейдите к Базы данных > Make_a_geoprocessing_model.gdb.
Выходные данные, CommercialCampgrounds_Buffer, были записаны в базу геоданных проекта по умолчанию. Вы можете отправить выходные данные модели в ее собственную базу геоданных, чтобы сохранить их отдельно от других наборов данных. Вы измените местоположение выходных данных модели позднее в этом учебном руководстве.
- В базе геоданных щелкните правой кнопкой CommercialCampgrounds_Buffer и щелкните Удалить . В окне запроса Удалить щелкните Да.

- Сделайте активным вид модели.
У инструмента и его переменной выходных данных появились тени. Это означает, что процесс уже был выполнен. Чтобы подготовить модель к повторному запуску, следует проверить ее.
- На вкладке ModelBuilder в группе Запустить щелкните Проверить .
Тени будут удалены, и модель станет готова к запуску. Вы присвоите модели имя и сохраните ее, а затем продолжите работу.
- На вкладке ModelBuilder в группе Модель щелкните Свойства .
- В диалоговом окне Свойства инструмента на вкладке Общие измените Имя на SummarizeInvasiveSpecies.
- Измените Подпись на Вычисление ареалов инвазивных видов.
- Нажмите OK.
- На вкладке ModelBuilder в группе Модель щелкните Сохранить .
Настройка модели как инструмента геообработки
Как вы видите, модель можно запустить из вида модели. Модель также можно запускать как инструмент геообработки. Это более распространенный и удобный способ запуска модели.
Это более распространенный и удобный способ запуска модели.
- На панели Каталог перейдите к Наборы инструментов > Make_a_geoprocessing_model.tbx > Summarize Invasive Species.
- На панели Каталог щелкните правой кнопкой модель Вычисление ареалов инвазивных видов и выберите Открыть.
- Сделайте активным вид модели.
Обратите внимание, что теперь вид модели называется Summarize Invasive Species.
- Щелкните правой кнопкой мыши переменную входных данных Commercial Campgrounds и выберите Параметр.
- На вкладке ModelBuilder в группе Модель щелкните Сохранить .
- На панели Каталог щелкните правой кнопкой инструмент модели Summarize Invasive Species и щелкните Открыть.
- Сделайте активным вид модели. Щелкните правой кнопкой на переменной входных данных Commercial Campgrounds и нажмите Переименовать. В поле редактирования измените имя на Местоположения контактов с человеком.
 Нажмите клавишу Enter.
Нажмите клавишу Enter.Это изменяет имя переменной в модели и имя соответствующего параметра в инструменте геообработки. (Вы не сможете увидеть изменения, пока заново не откроете инструмент.)
- Щелкните правой кнопкой мыши элемент инструмента Буфер и выберите Создать переменную > Из параметра > Расстояние [значение или поле].
Новый элемент представлен другим оттенком синего цвета, поскольку это переменная значения, а не переменная данных.
- Если необходимо, передвиньте выделенную переменную Расстояние [значение или поле] так, чтобы она не перекрывала другие элементы модели.
- Щелкните правой кнопкой переменную Расстояние [значение или поле] и выберите Параметр. Щелкните свободное место в виде модели, чтобы снять выделение переменной.
- Щелкните правой кнопкой мыши значение переменной Расстояние [значение или поле] и выберите Переименовать. В поле редактирования измените имя на Буферное расстояние.
 Нажмите клавишу Enter.
Нажмите клавишу Enter. - Щелкните правой кнопкой на переменной выходных данных Commercial_Campgrounds_Buffer и выберите Переименовать. Измените имя на Contact Location Buffers и нажмите клавишу Enter.
- На вкладке ModelBuilder в группе Модель щелкните Сохранить .
- На панели Каталог щелкните правой кнопкой инструмент модели Summarize Invasive Species в наборе инструментов Make_a_geoprocessing_model и щелкните Открыть.
- Сделайте активным вид модели. Правой кнопкой мыши щелкните переменную данных Местоположения контактов с человеком и выберите Открыть.
- Выделите значение по умолчанию Commercial Campgrounds и нажмите клавишу Удалить. Нажмите OK.
Процесс модели станет серым (модель не готова к запуску), так как вы удалили заданное входное значение инструмента Буфер. Что ожидаемо. Модель в таком состоянии нельзя запустить из вида модели, но ее можно запустить в качестве инструмента геообработки при заполнении его параметров.

- Щелкните правой кнопкой мыши значение переменной Буферное расстояние и выберите Открыть.
- Выделите значение расстояния по умолчанию 5 и нажмите клавишу Удалить. Нажмите OK.
Все элементы модели станут серыми.
- На вкладке ModelBuilder в группе Модель щелкните Сохранить .
- На панели Каталог щелкните правой кнопкой инструмент Summarize Invasive Species и щелкните Открыть.
Завершение построения модели
Чтобы завершить построение модели, вы добавите переменную входных данных инвазивных видов и инструмент Суммировать в пределах. Вы соедините входные переменные с инструментами и зададите параметры модели.
Ранее вы создавали переменную входных данных перетаскивая слой с панели Содержание в вид модели. Теперь вы создадите переменную с помощью ленты.
- Сделайте активным вид модели. На вкладке ModelBuilder щелкните группу Вставить, а затем – Переменная .

- В диалоговом окне Тип данных переменной щелкните на ниспадающей стрелке и выберите Векторный слой. (Может понадобиться прокрутить список вверх). Щелкните ОК.
- Перетащите выбранную переменную в пустую часть модели.
- Переименуйте переменную данных Слой объектов в Invasive Species.
- Сделайте переменную Invasive Species параметром модели.
При запуске модели как инструмента геообработки у вас будет возможность выбрать, какие инвазивные виды будут проанализированы.
- На вкладке ModelBuilder щелкните группу Вставить, а затем – Инструменты .
- На панели Геообработка в поле поиска введите Суммировать в пределах.
- Перетащите инструмент Суммировать в пределах с панели Геообработка в вид модели.
- Наведите курсор на переменную данных Инвазивные виды. Перетащите линию соединения к инструменту Суммировать в пределах. Отпустите кнопку мыши и нажмите Ввести объекты суммирования во всплывающем меню.

- Соедините переменную данных Contact Location Buffers с инструментом Суммировать в пределах, чтобы задать Входные полигоны.
Позже в этом разделе вы настроите компоновку модели.
- Щелкните правой кнопкой мыши переменную данных Выходной класс пространственных объектов и выберите Параметр.
Настройка этой переменной в виде параметра модели позволяет указать имя для выходного класса объектов и изменить выходное местоположение при запуске инструмента.
- Щелкните снова правой кнопкой мыши на переменной данных Выходной класс объектов и затем Открыть.
- Выделите полностью путь Выходной класс объектов и нажмите клавшиу Delete. Нажмите OK.
- Щелкните правой кнопкой инструмент Суммировать в пределах и выберите Создать переменную > Из параметра > Единицы измерения объекта.
- Перетащите выбранную переменную на свободную часть модели, если необходимо.
- Переименуйте значение Единицы измерения объекта в Площадные единицы измерения.

Этот параметр позволяет выбрать единицы измерения, такие как гектары, в которых будут суммироваться площади, занятые инвазивными видами.
- Сделайте переменную Area Unit of Measure параметром модели.
- Снимите выделение с переменной Площадные единицы измерения.
- На вкладке ModelBuilder в группе Вид щелкните Автокомпоновка .
- По желанию вы можете выбрать элементы модели и переместить их в лучшие местоположения.
- Щелкните Сохранить , чтобы сохранить модель.
- На панели Каталога откройте инструмент Вычисление ареалов инвазивных видов.
Задание свойств и параметров среды модели
Перед запуском модели вы измените порядок следования параметров в инструменте геообработки. Также вы зададите символы, используемые по умолчанию для выходных данных. Наконец, вы сохраните выходные данные модели в собственную базу геоданных.
- На вкладке ModelBuilder в группе Модель щелкните Свойства .

- В диалоговом окне Свойства инструмента перейдите на вкладку Параметры.
Порядок параметров сверху вниз в диалоговом окне Параметры инструмента соответствуют порядку, в котором они следуют в инструменте геообработки. Этот порядок отражает последовательность, в которой вы задавали переменные в качестве параметров модели.
- В первом столбце таблицы (столбец с номерами) нажмите цифру 4, чтобы выбрать последнюю строку, которая представляет собой параметр Единицы измерения площади.
- Перетащите выбранный параметр на одну позицию выше параметра Выходной класс обеъктов.
При перетаскивании параметра, его положение будет обозначаться горизонтальной чертой серого цвета.
- Пролистайте таблицу, чтобы увидеть другие столбцы.
- В последней строке таблицы с параметром Выходной класс объектов щелкните ячейку в столбце Символы таблицы и нажмите на появившуюся кнопку Обзор .
- В окне обзора, в разделе Проект, щелкните Папки.

- В смежном окне дважды щелкните Make_a_geoprocessing_model_1 > commondata > userdata.
- Щелкните файл BufferSymbol.lyrx, чтобы выбрать. И нажмите OK.
Путь к файлу слою появляется в столбце Символы таблицы параметров.
- Щелкните OK в диалоговом окне Свойства инструмента.
- Щелкните Сохранить , чтобы сохранить модель.
Ранее вы видели, что ваши выходные данные модели сохранялись в базу геоданных по умолчанию. Эта база геоданных также содержит входные данные модели, например, классы объектов инвазивных видов растений, кемпингов, дорог и троп. Чтобы сохранить выходные данные модели отдельно от входных данных, вы создадите новую базу геоданных и сделаете ее базой геоданных по умолчанию.
- На панели Каталог щелкните правой кнопкой Базы данных и выберите Новая файловая база геоданных .
- В текстовом поле Имя введите Model_output.
- Щелкните Сохранить.

- На панели Каталог при необходимости разверните Базы данных, чтобы увидеть новую базу геоданных.
Теперь вам надо поместить выходные данные в эту базе геоданных.
- На панели Каталог щелкните правой кнопкой мыши на базе геоданных Model_output и затем Сделать по умолчанию .
На базе геоданных появится значок, который говорит о том, что это теперь база геоданных проекта по умолчанию. Результаты геообработки будут сохраняться в этой базе геоданных, если не будет задано иное.
- На панели Каталога откройте инструмент Вычисление ареалов инвазивных видов.
В нижней части панели инструмента появится параметр Выходной класс объектов. Инструмент готов к использованию.
- Закройте вид модели. Убедитесь, что активен вид карты Marlborough.
Запуск инструмента Вычисление ареалов инвазивных видов
Вы запустите инструмент и проверите выходные данные.
- На панели Содержание отключите слои Commercial Campgrounds и Nassella Tussock Range.

- Включите слои Campsites и White Edged Nightshade Range.
На карте, на севере региона один или два кемпинга выглядят расположенными поблизости от мест распространения паслена окаймленного.
- На панели Геообработка в инструменте Summarize Invasive Species задайте для параметра Human Contact Locations значение Campsites.
- Задайте Буферное расстояние 1.5 километра.
- Задайте для Invasive Species значение White Edged Nightshade Range.
- Задайте для Единицы измерения площади значение ГЕКТАРЫ.
- В поле имени файла Выходного класса объектов введите Nightshade_Near_Campsites.
- Щелкните Запустить.
- Увеличьте карту, чтобы увидеть буферы.
- На панели Содержание щелкните правой кнопкой мыши слой Nightshade_Near_Campsites и щелкните Таблица атрибутов .
- Если необходимо, пролистайте таблицу.
 Щелкните правой кнопкой заголовок поля Summarized area in HECTARES и выберите Сортировать по убыванию .
Щелкните правой кнопкой заголовок поля Summarized area in HECTARES и выберите Сортировать по убыванию .Только в одном кемпинге в радиусе 1.5 км есть белоснежный паслен. Значение суммированной площади равно 7,35 га.
- Закройте таблицу.
- На панели Каталог перейдите к Базы данных > Model_output.gdb и убедитесь, что Nightshade_Near_Campsites был сохранен в эту базу геоданных.
- Самостоятельно воспользуйтесь инструментом Вычисление ареалов инвазивных видов, чтобы проанализировать другие отношения между инвазивными видами и местами возможного контакта с человеком.
Документирование инструмента Вычисление ареалов инвазивных видов
Ваша последняя задача – задокументировать инструмент геообработки. Документация будет выглядеть как справка к другим инструментам геообработки: в ней описывается назначение инструмента и его использование.
- На панели Геообработка, в инструменте Вычисление ареалов инвазивных видов щелкните Справка , чтобы открыть справку инструмента.

Справка инструмента открывается в окне или вкладке браузера. (URL указывает на локальный файл на компьютере.) У инструмента есть заголовок, но никакой другой информации. Для создания документации следует отредактировать метаданные инструмента.
- Закройте вкладку или окно браузера со справкой инструмента.
- На панели Каталог щелкните правой кнопкой инструмент Summarize Invasive Species и щелкните Редактировать метаданные .
- В виде метаданных в окне Теги введите или скопируйте и вставьте следующие теги: суммировать, биобезопасность, инвазивные виды, Новая Зеландия.
Проверьте, что теги разделены запятыми. Теги являются ключевыми словами, которые помогают пользователям найти инструмент, если он опубликован как пакет геообработки или часть пакета проекта.
- В поле Краткая информация (резюме) введите следующий текст: Этот инструмент предназначен для анализа проблем биобезопасности растений в Новой Зеландии.
 Он суммирует ареалы инвазивных видов трав около местоположений, где человек может входить в контакт с растениями и способствовать распространению их семян.
Он суммирует ареалы инвазивных видов трав около местоположений, где человек может входить в контакт с растениями и способствовать распространению их семян. - В поле Использование введите следующий текст:
- В разделе Синтаксис разверните Местоположения_контактов_с_человеком. В поле Пояснение к диалоговому окну введите следующий текст: Объекты представляют собой местоположения, где человек может контактировать с инвазивными видами растений.
- В разделе Синтаксис разверните Буферное_расстояние. В поле Пояснение к диалоговому окну введите следующий текст: Расстояние, определяющее зоны вокруг местоположений возможного контакта человека с растениями, которые будут анализироваться на наличие инвазивных видов.
- В разделе Синтаксис разверните Инвазивные_виды. В поле Пояснение к диалоговому окну введите следующий текст: Инвазивные виды растений, анализируемые на близость к местоположениям, где человек может контактировать с ними.

- В разделе Синтаксис разверните Площадные_единицы_измерения. В поле Пояснение к диалоговому окну введите следующий текст: Единицы измерения площади ареала инвазивных видов в пределах буферной зоны.
- В разделе Синтаксис разверните Выходной_класс_пространственных_объектов. В поле Пояснение к диалоговому окну введите следующий текст: Имя выходного класса объектов.
- На ленте во вкладке Метаданные в группе Управление метаданными нажмите Сохранить .
- На панели Каталог щелкните правой кнопкой инструмент Summarize Invasive Species и щелкните Открыть.
- На панели Геообработка наведите курсор на Справку , чтобы увидеть краткое описание инструмента.
- Наведите курсор на значок Информация, который появляется рядом с параметрами инструмента, чтобы увидеть свои объяснения.
- Закройте вид метаданных Summarize Invasive Species.
- На панели Инструменты быстрого доступа щелкните Сохранить , чтобы сохранить проект.

Эта модель геообработки автоматизирует рабочий процесс анализа. Он может использоваться для анализа и комбинирования с информацией о местоположениях контакта с людьми, инвазивных видах или отношений расстояния. Если вы хотите поделиться своей моделью с другими пользователями, они смогут запустить инструмент геообработки, не открывая вид модели.
Модель может быть опубликована несколькими разными способами. Если вы публикуете ее как пакет проекта (файл .ppkx), пакет будет содержать модель, вид карты и все данные проекта. Если вы публикуете ее как пакет геообработки (файл .gptx), пакет будет содержать только инструмент геообработки и примеры слоев входных и выходных данных. Если вы планируете опубликовать свою модель, возможно следует заменить встроенными переменными жестко заданные пути к входным и выходным наборам данных.
Другие учебные руководства, использующие расширенные функции ModelBuilder, доступны в справочной системе. См., например, Работа с Итерированием классов объектов.
Связанные разделы
Отзыв по этому разделу?
Модели Django · HonKit
Нам нужно что-то, что будет хранить все записи нашего блога. Но прежде давай поговорим о вещах, называемых объектами.
Объекты
В программировании существует особая концепция, она называется объектно-ориентированным программированием. Идея заключается в том, что вместо скучной последовательности инструкций мы моделируем вещи и описываем, как они взаимодействуют друг с другом.
Так что же такое объект? Это совокупность поведения и свойств. Звучит странно, но мы приведем пример.
Если мы хотим смоделировать кошку, то создадим объект Cat, который обладает определенными свойствами, например, color (цвет), age (возраст), mood (настроение: плохое, хорошее, сонное ;)), owner (хозяин, например, другой объект — Person — или, если кошка дикая, это свойство будет пустым).
Объект Cat будет иметь набор определённых действий: purr (мурчать), scratch (царапаться) или feed (кормить, где мы дадим кошке немного CatFood — кошачьей еды, которая так же может быть отдельным объектом со своими свойствами, например, taste — вкусом).
Cat -------- color age mood owner purr() scratch() feed(cat_food)
CatFood -------- taste
Основная идея, таким образом, заключается в описании объекта в коде, используя его параметры (свойства объекта) и доступные ему действия (методы).
Так как же мы смоделируем запись в блоге? Нам же нужен блог, верно?
Для начала стоит ответить на вопрос: что такое запись в блоге? Какие свойства она имеет?
Ну, запись наверняка содержит какой-то текст и заголовок, верно? Было бы неплохо также знать, кто её написал — так что нам нужен автор. Ну и в заключение, нам нужно знать, когда запись создана и когда опубликована.
Post -------- title text author created_date published_date
Какие вещи можно сделать с записью в блоге? Было бы неплохо иметь метод для её публикации, согласна?
Так что нам пригодится метод publish.
Ну и раз уж мы определились с тем, что хотим получить, давай начнем моделирование в Django!
Модель в Django
Зная, что представляет из себя объект, мы можем создать модель Django для записи в блоге.
Модель в Django — это объект определённого свойства: он хранится в базе данных. База данных представляет собой совокупность различных данных. Это то место, где ты будешь хранить информацию о своих пользователях, записях в блоге и т.д. Мы будем использовать базу данных SQLite для хранения информации. Это стандартная база данных в Django — её сейчас вполне хватит для наших нужд.
Ты можешь представить модель в базе данных как электронную таблицу с колонками (полями) и строками (данными).
Создание приложения
Для аккуратности мы создадим отдельное приложение в нашем проекте. Очень удобно иметь хорошо организованное рабочее место с самого начала. Для создания приложения нам понадобится набрать следующую инструкцию в командной строке (из директории
Очень удобно иметь хорошо организованное рабочее место с самого начала. Для создания приложения нам понадобится набрать следующую инструкцию в командной строке (из директории djangogirls, где находится файл manage.py):
Mac OS X и Linux:
(myvenv) ~/djangogirls$ python manage.py startapp blog
Windows:
(myvenv) C:\Users\Name\djangogirls> python manage.py startapp blog
Обрати внимание, что в нашем проекте появилась новая папка blog, которая содержит некоторые файлы. Таким образом, структура нашего проекта будет выглядеть так:
djangogirls
├── blog
│ ├── __init__.py
│ ├── admin.py
│ ├── apps.py
│ ├── migrations
│ │ └── __init__.py
│ ├── models.py
│ ├── tests.py
│ └── views.py
├── db.sqlite3
├── manage.py
└── mysite
├── __init__.py
├── settings.py
├── urls.py
└── wsgi.py
После того, как приложение создано, нам нужно сообщить Django, что теперь он должен его использовать. Мы сделаем это с помощью файла
Мы сделаем это с помощью файла mysite/settings.py. Нам нужно найти INSTALLED_APPS и добавить к списку 'blog', прямо перед ]. Конечный результат должен выглядеть следующим образом:
mysite/settings.py
INSTALLED_APPS = [
'django.contrib.admin',
'django.contrib.auth',
'django.contrib.contenttypes',
'django.contrib.sessions',
'django.contrib.messages',
'django.contrib.staticfiles',
'blog',
]
Создание модели записи в блоге
В файле blog/models.py мы определяем все Модели — модель записи для блога также пойдёт сюда.
Открой файл blog/models.py, удали весь текст и вставь на его место следующий код:
blog/models.py
from django.conf import settings
from django.db import models
from django.utils import timezone
class Post(models.Model):
author = models.ForeignKey(settings.AUTH_USER_MODEL, on_delete=models.CASCADE)
title = models. CharField(max_length=200)
text = models.TextField()
created_date = models.DateTimeField(default=timezone.now)
published_date = models.DateTimeField(blank=True, null=True)
def publish(self):
self.published_date = timezone.now()
self.save()
def __str__(self):
return self.title
CharField(max_length=200)
text = models.TextField()
created_date = models.DateTimeField(default=timezone.now)
published_date = models.DateTimeField(blank=True, null=True)
def publish(self):
self.published_date = timezone.now()
self.save()
def __str__(self):
return self.title
Убедись, что использовала два символа нижнего подчёркивания (
_) с обеих сторон от методаstr. Это соглашение часто используется при программировании на Python, и иногда его называют «dunder» (сокращение от англ. «double-underscore»).
Смотрится страшно, да? Но не волнуйся, мы объясним, что значит каждая строка кода!
Строки, начинающиеся с from или import, открывают доступ к коду из других файлов. Так что вместо того, чтобы копировать и вставлять один и тот же код во все файлы, ты можешь сослаться на него при помощи from ... import ....
class Post(models. — эта строка определяет нашу модель ( Model):
Model):объект).
class— это специальное ключевое слово для определения объектов.Post— это имя нашей модели, мы можем поменять его при желании (специальные знаки и пробелы использовать нельзя). Всегда начинай имена классов с прописной буквы.models.Modelозначает, что объект Post является моделью Django, так Django поймет, что он должен сохранить его в базу данных.
Дальше мы задаем свойства, о которых уже говорили: title, text, created_date, published_date и author. Чтобы это сделать, нам нужно определиться с типом полей (это текст? число? дата? ссылка на другой объект? например, на пользователя?).
models.CharField— так мы определяем текстовое поле с ограничением на количество символов.models.TextField— так определяется поле для неограниченно длинного текста. Выглядит подходящим для содержимого поста, верно?
Выглядит подходящим для содержимого поста, верно?models.DateTimeField— дата и время.models.ForeignKey— ссылка на другую модель.
Мы не будем объяснять каждую запятую, поскольку на это уйдет слишком много времени. Ознакомься с официальной документаций Django: если хочешь узнать больше о полях моделей и о том, как определять разные объекты, то эта ссылка может помочь: https://docs.djangoproject.com/en/1.11/ref/models/fields/#field-types.
Что насчёт def publish(self):? Это как раз метод публикации для записи, о котором мы говорили. def означает, что создаётся функция/метод, а publish — это название этого метода. Можно изменить имя метода, если хочешь. Существует правило для имён функций: нужно использовать строчные буквы, а пробелы заменять на подчёркивания. Например, метод, вычисляющий среднюю цену, может называться calculate_average_price.
Методы часто возвращают что-то. Например, метод
Например, метод __str__. В нашем случае после вызова метода __str__() мы получим текст (строку) с заголовком записи.
Также обрати внимание, что оба метода def publish(self): и def __str__(self): внутри класса имеют дополнительный отступ. Поскольку в Python важны отступы, нам необходимо использовать их для методов внутри класса — иначе методы не будут принадлежать к классу, и при запуске программы может получиться что-то неожиданное.
Если тема моделей тебе до сих пор непонятна — не стесняйся обратиться к тренеру! Мы знаем, что она действительно сложна, особенно когда ты изучаешь параллельно объекты и функции. Но мы надеемся, что всё это больше не кажется тебе магией!
Создаём таблицы моделей в базе данных
Последним шагом будет добавление нашей модели в базу данных. Сначала мы должны дать Django знать, что сделали изменения в нашей модели (мы её только что создали!). Набери python manage.. Должно получиться примерно так: py makemigrations blog
py makemigrations blog
command-line
(myvenv) ~/djangogirls$ python manage.py makemigrations blog Migrations for 'blog': blog/migrations/0001_initial.py: - Create model Post
Примечание: не забудь сохранить отредактированные файлы. Иначе твой компьютер выполнит команду с их предыдущей версией, и могут появиться неожиданные ошибки.
Django создал для нас файл с миграцией для базы данных. Набери python manage.py migrate blog, результат должен быть следующим:
command-line
(myvenv) ~/djangogirls$ python manage.py migrate blog Operations to perform: Apply all migrations: blog Running migrations: Rendering model states... DONE Applying blog.0001_initial... OK
Ура! Модель записи для блога теперь в базе данных, и было бы неплохо посмотреть на неё, верно? Тогда переходи к следующей главе!
Что такое модель создания? – Core Academy of Science
Весной 1900 года группа ныряльщиков за губками, возвращавшаяся домой на греческие острова, случайно укрылась от шторма у северо-восточного побережья крошечного острова Антикифера. После шторма водолазы решили исследовать морское дно под собой в надежде найти губки в неизведанных водах. Вместо этого они нашли одно из самых богатых и знаменитых кораблекрушений Древней Греции. Их первоначальные исследования нашли бронзовую руку, оторванную от статуи, а последующие исследования обнаружили на поверхности множество знаменитых статуй из бронзы и мрамора. Вместе с этими произведениями искусства прибыло много небольших фрагментов инкрустированной бронзы, которые считались осколками разбитых статуй, которые можно было использовать при их реконструкции. Исследования, проведенные под руководством Жака Кусто более 75 лет спустя, позволили обнаружить монеты из затонувшего корабля, датированные серединой первого века до нашей эры. Обломкам и артефактам более 2000 лет.
После шторма водолазы решили исследовать морское дно под собой в надежде найти губки в неизведанных водах. Вместо этого они нашли одно из самых богатых и знаменитых кораблекрушений Древней Греции. Их первоначальные исследования нашли бронзовую руку, оторванную от статуи, а последующие исследования обнаружили на поверхности множество знаменитых статуй из бронзы и мрамора. Вместе с этими произведениями искусства прибыло много небольших фрагментов инкрустированной бронзы, которые считались осколками разбитых статуй, которые можно было использовать при их реконструкции. Исследования, проведенные под руководством Жака Кусто более 75 лет спустя, позволили обнаружить монеты из затонувшего корабля, датированные серединой первого века до нашей эры. Обломкам и артефактам более 2000 лет.
Через два года после обнаружения обломков, в мае 1902 года, работники Национального археологического музея в Афинах наткнулись на один из лишних кусочков ржавой бронзы и заметили, что на нем была надпись и что-то похожее на колеса. Этот странный набор фрагментов стал известен как «Антикиферский механизм». Ранние мнения о значении устройства разделились между теми, кто полагал, что это астролябия, относительно простой инструмент, используемый для измерения высоты астрономического объекта над горизонтом, и теми, кто полагал, что это должно быть что-то гораздо более сложное.
Этот странный набор фрагментов стал известен как «Антикиферский механизм». Ранние мнения о значении устройства разделились между теми, кто полагал, что это астролябия, относительно простой инструмент, используемый для измерения высоты астрономического объекта над горизонтом, и теми, кто полагал, что это должно быть что-то гораздо более сложное.
Последующие исследования показали, что Антикиферский механизм представлял собой сложный механический компьютер, используемый для расчета положения планет на небе, взаимосвязи между лунными фазами и солнечным годом и даже времени затмений. Сложность Механизма беспрецедентна для того времени, настолько, что некоторые люди предположили, что на самом деле он не так стар, как кораблекрушение. Тем не менее, математические детали моделируемой им геоцентрической Вселенной прочно помещают ее в мир древнегреческой астрономии.
Спустя более 1300 лет после того, как этот Механизм был утерян у берегов Антикитеры, внимание Западной Европы привлекли еще два знаменитых астрономических часа. В Англии Ричард Валлингфорд изобрел устройство, которое показывало время, движение солнца и луны и приливы и отливы. В городе Падуя, ныне на севере Италии, Джованни де Донди потратил шестнадцать лет на то, чтобы построить такие же часы, которые отслеживали движение планет. Эти устройства были настолько знамениты, что представление о Вселенной как о больших часах укоренилось в европейском воображении. В течение четырех столетий после изобретения этих двух часов Кеплером и Ньютоном были разработаны законы движения планет и гравитации, которые подтвердили регулярность Вселенной, но идея космоса с часовым механизмом уходит своими корнями в Средневековье. Возраст с этими часами.
В Англии Ричард Валлингфорд изобрел устройство, которое показывало время, движение солнца и луны и приливы и отливы. В городе Падуя, ныне на севере Италии, Джованни де Донди потратил шестнадцать лет на то, чтобы построить такие же часы, которые отслеживали движение планет. Эти устройства были настолько знамениты, что представление о Вселенной как о больших часах укоренилось в европейском воображении. В течение четырех столетий после изобретения этих двух часов Кеплером и Ньютоном были разработаны законы движения планет и гравитации, которые подтвердили регулярность Вселенной, но идея космоса с часовым механизмом уходит своими корнями в Средневековье. Возраст с этими часами.
Несмотря на столетия, разделяющие их изобретение, Антикитерский механизм и эти средневековые часы пытаются сделать одно и то же. Все они модели, в данном случае миниатюрные устройства, которые представляют собой нечто гораздо большее. Модели могут быть очень простыми и репрезентативными, например, статуи или картины, где фиксируются и отображаются только поверхностные детали. Такие модели, как эти компьютеры с редуктором, пытаются изобразить более сложные характеристики движения и времени. Говоря современным языком, это научные модели.
Такие модели, как эти компьютеры с редуктором, пытаются изобразить более сложные характеристики движения и времени. Говоря современным языком, это научные модели.
Антикиферский механизм был сделан из бронзы и дерева, но современные научные модели чаще всего носят чисто теоретический характер. Они существуют в мире математики, компьютерных программ и письменной речи ученых. Хотя теоретически они не менее убедительны и мощны, чем физические часы. Скорее всего, вы регулярно взаимодействуете с научными моделями и полагаетесь на них, даже если не знаете об этом. Научные модели лежат в основе прогнозов погоды, медицинских решений и практически любой технологии, с которой вы сталкиваетесь.
Цель научной модели — запечатлеть детали окружающего нас мира. Научные модели строятся на наблюдениях и закономерностях Божьего творения. Модели выходят за рамки просто научных законов. Например, закон всемирного тяготения гласит, что сила притяжения между двумя телами прямо пропорциональна произведению их масс и обратно пропорциональна квадрату расстояния между ними. Звучит интересно, не так ли? Причина, по которой люди так увлечены законом гравитации, заключается в том, что он позволяет нам делать: Планеты в будущем можно было предсказать. Все научные модели работают одинаково, объединяя наблюдения и закономерности (законы) в представления реальности.
Звучит интересно, не так ли? Причина, по которой люди так увлечены законом гравитации, заключается в том, что он позволяет нам делать: Планеты в будущем можно было предсказать. Все научные модели работают одинаково, объединяя наблюдения и закономерности (законы) в представления реальности.
Научные модели могут быть хорошими или плохими, в зависимости от деталей модели, но все «хорошие» и «плохие» модели зависят от того, что вы от них ожидаете. Даже неправильные модели могут точно объяснить наблюдения. Антикиферский механизм плохо представляет то, что мы знаем о космосе, поскольку он помещает солнце на орбиту вокруг Земли, но, тем не менее, он вычисляет календари и лунные фазы с приличной точностью. Как ни странно, даже «неправильные» модели могут быть точными.
Модель сотворения, насколько я понимаю, представляет собой научную модель, построенную с использованием двух типов данных: информации, полученной в результате наблюдения за творением Бога, и информации, полученной в результате изучения Божьего откровения. Модель творения синтезирует все в грандиозное представление о творении рук Бога. В некотором смысле модель сотворения — это своего рода проповедь или гимн, что-то, что мы сочиняем или произносим в ответ на Божью природу или работу в нашей жизни. Мы видим, что сделал Бог, и мы хотим понять это и провозгласить эту работу другим.
Модель творения синтезирует все в грандиозное представление о творении рук Бога. В некотором смысле модель сотворения — это своего рода проповедь или гимн, что-то, что мы сочиняем или произносим в ответ на Божью природу или работу в нашей жизни. Мы видим, что сделал Бог, и мы хотим понять это и провозгласить эту работу другим.
Вот почему мы, ученые-креационисты, создаем модели. Мы мотивированы открывать дела Божьи. Мы с нетерпением ждем новых открытий, и мы постоянно работаем над нашим пониманием и улучшаем различные части наших моделей. Со временем наши модели лучше изображают Божье творение. Сто лет назад лишь горстка креационистов активно интересовалась вопросами науки и происхождения. Геолог-самоучка Джордж Маккриди Прайс учил, что летопись окаменелостей на самом деле представляла собой беспорядочную смесь слоев, где можно было найти любой слой поверх любого другого слоя. Сегодня наши геологические модели Потопа объясняют мелкие детали образования горных пород, а также включают более крупные свидетельства движения континентов. Мы прошли долгий путь.
Мы прошли долгий путь.
Тем не менее, научные модели никогда не создаются, по крайней мере, в этой жизни. Точно так же, как мы не всегда понимаем Божью работу в нашей жизни, мы не всегда понимаем детали Божьего творения. Как летопись окаменелостей стала такой упорядоченной? Как мы можем видеть звезды так далеко? Кем были неандертальцы? Есть еще много вопросов, на которые нужно ответить, и еще больше творения, которое нужно исследовать.
Мы прошли долгий путь с момента создания Антикитерского механизма. Куда мы пойдем завтра? И, возможно, что еще более важно, как вы можете участвовать в провозглашении великих и могущественных дел Бога? Вот несколько предложений.
- Просите Бога благословить тех, кто участвует в этой работе, и студентов, стремящихся принять участие.
- Рассмотрите возможность финансового взноса в Стипендиальный фонд Сандерса для поддержки студенческих исследований.
- Знаете кого-нибудь с пытливым умом и способностями к науке? Они могут быть просто лидерами науки о сотворении завтрашнего дня.
 Поощряйте их участие.
Поощряйте их участие. - Возможно, у вас есть собственные способности и хороший научный опыт, но вам просто интересно, как принять участие. Я рекомендую прочитать мою книгу Квест за хорошие идеи по открытым вопросам.
Модели — замечательные вещи, и мне посчастливилось участвовать в моделировании Божьего творения. Теперь у меня есть привилегия призывать других присоединиться к этой работе. Я надеюсь, вы присоединитесь к этому величайшему приключению, пока мы вместе исследуем Божье творение.
(Фото Антикиферского механизма, Тилемахос Эфтимиадис, wikimedia)
5 Основные этапы процесса
5 Основные этапы процесса
Создание 3D-моделей — одна из самых популярных услуг компьютерной графики, используемая в архитектуре, дизайне интерьеров и продуктов, а также в автомобильной и игровой индустрии.
Что касается производителей, то профессиональное 3D моделирование компания предоставляет массу возможностей для их бизнеса. Он обеспечивает быстрое и удобное прототипирование, качественные решения для анимации, а также первоклассную 3D-визуализацию для различных целей — каталогов, списков, онлайн- и офлайн-рекламы, социальных сетей и т. д. Честно говоря, сегодня ни одна маркетинговая кампания не может обойтись без челюстно-лицевой рекламы. сбрасывание визуализаций и высококачественных 3D-моделей. Ведь современные проблемы требуют современных решений!
Он обеспечивает быстрое и удобное прототипирование, качественные решения для анимации, а также первоклассную 3D-визуализацию для различных целей — каталогов, списков, онлайн- и офлайн-рекламы, социальных сетей и т. д. Честно говоря, сегодня ни одна маркетинговая кампания не может обойтись без челюстно-лицевой рекламы. сбрасывание визуализаций и высококачественных 3D-моделей. Ведь современные проблемы требуют современных решений!
Рынок 3D визуализации насыщен предложениями как от фрилансеров, так и от компьютерных студий. Но все подрядчики называют разные цены. Чтобы по-настоящему разобраться в ситуации и сделать правильный выбор, маркетологам и производителям в первую очередь необходимо понять, как происходит создание 3D-моделей. Немного углубившись в тему, они смогут понять, за что платят за создание 3D-моделей, а затем выбрать лучшего подрядчика. Итак, начнем, 5 основных этапов создания 3D модели.
№1. Постановка точной задачи
Во-первых, маркетологи и производители должны точно знать, каковы их цели с точки зрения создания 3D-моделей. Тип и сложность 3D-объекта зависят от его будущего использования, будь то статичная красивая визуализация или решение движения — анимация, 360-градусный обзор продукта, виртуальная реальность и так далее.
Тип и сложность 3D-объекта зависят от его будущего использования, будь то статичная красивая визуализация или решение движения — анимация, 360-градусный обзор продукта, виртуальная реальность и так далее.
Итак, клиент ставит перед студией компьютерной графики четкую задачу, отправляя им развернутый бриф с образцами, референсами и чертежами продукции. После этого вместе с руководителем проекта студии они могут обсудить и уточнить все детали, а также назначить сроки создания 3D-модели.
Профессиональные 3D-студии знают, насколько важен этот этап. Имея большой опыт в 3D-моделировании, они могут подсказать, какая 3D-модель для каких целей лучше, разбить процесс на этапы и распределить задачи между экспертами в команде. В результате, чем яснее первоначальный бриф, тем быстрее идет процесс и требуется меньше корректировок.
№2. Построение базовой геометрии
Существуют различные способы создания 3D-модели с использованием твердотельных, поверхностных и каркасных типов 3D-моделирования. Solid опирается на создание нового объекта путем преобразования базовых геометрических форм — кубов, шаров, цилиндров и т. д. Поверхностный метод основан на рисовании направляющих линий, которые создают поверхность. А под полигональным моделированием подразумевается создание 3D модели из полигонов. В основном, это самый популярный вид 3D-моделирования для проектирования мебели.
Solid опирается на создание нового объекта путем преобразования базовых геометрических форм — кубов, шаров, цилиндров и т. д. Поверхностный метод основан на рисовании направляющих линий, которые создают поверхность. А под полигональным моделированием подразумевается создание 3D модели из полигонов. В основном, это самый популярный вид 3D-моделирования для проектирования мебели.
Многоугольник состоит из трех точек-вершин, объединенных общими ребрами. Его основная структура треугольная, и эти треугольники могут быть объединены в более сложные поверхности. Чем больше полигонов имеет созданный 3D-объект, тем ровнее его поверхность и качественнее. Но сама такая 3D-модель становится более «тяжелой». «Тяжелый» в этом контексте означает, что для рендеринга требуется много ресурсов. Поэтому он не работает для приложений VR, AR и видеоигр. Тем не менее, это создание 3D-модели идеально подходит для красивых фотореалистичные 3D визуализации .
№3. Настройка полигонов и топологии
Часто после создания базовой геометрии 3D-модели необходимо настроить полигоны в соответствии с формой объекта. Это жизненно важно для тех типов 3D-объектов, которые могут быть слишком тяжелыми для запуска в приложениях, VR, AR или компьютерных играх, созданных на движке Unity. В этих случаях такие процессы, как топология и ретопология, могут решить проблему за счет уменьшения количества полигонов. Если меньшие полигоны точно повторяют форму и линии структуры, то 3D-модель становится легче, но сохраняет хорошее качество.
Это жизненно важно для тех типов 3D-объектов, которые могут быть слишком тяжелыми для запуска в приложениях, VR, AR или компьютерных играх, созданных на движке Unity. В этих случаях такие процессы, как топология и ретопология, могут решить проблему за счет уменьшения количества полигонов. Если меньшие полигоны точно повторяют форму и линии структуры, то 3D-модель становится легче, но сохраняет хорошее качество.
Топология и ретопология для создания 3D-моделей также позволяют уменьшить системные ошибки и другие виды ошибок. После их использования 3D-объекты не требуют больших ресурсов компьютера или смартфона, но передают изображение с высоким качеством и реалистичностью.
№4. Выбор материалов и текстур
Существует множество различных библиотек материалов для 3D-программ, где вы можете найти любые материалы и текстуры, начиная с камня, дерева, металлов, тканей и т. д.
Материалы определяют такие характеристики объекта, как прозрачность , твердость, отражательная способность и так далее. Однако они не имеют ни цвета, ни рисунка. А текстура — это, по сути, 2D-изображение поверхности, которая не имеет каких-либо ранее упомянутых свойств материала. Вместе они дают при создании 3D-модели как рисунок, так и рельеф поверхности.
Однако они не имеют ни цвета, ни рисунка. А текстура — это, по сути, 2D-изображение поверхности, которая не имеет каких-либо ранее упомянутых свойств материала. Вместе они дают при создании 3D-модели как рисунок, так и рельеф поверхности.
Обычно 3D художники используют готовые текстуры, но всегда могут создать свои по желанию клиента. Этот вариант идеально подходит для эксклюзивных материалов для нестандартных изделий с уникальным дизайном. Пользовательская текстура является гарантией того, что ни один другой 3D-художник не будет использовать ее для создания своей 3D-модели. Конечно, создание собственной текстуры требует времени и денег, но так опытный 3D-специалист сможет сделать именно тот материал, который нужен производителю.
№5. Отображение и применение текстур
Картографирование — это процесс создания текстурной карты и ее наложения на 3D-модель. Хотя 3D-объект представляет собой 3D-объем, карты всегда создаются в 2D. Это значит, что они представляют собой не более чем цветную плоскую картинку без рельефа поверхности. Поэтому, даже имея на руках различные карты, 3D-художникам приходится прикладывать усилия, чтобы текстура выглядела реалистично.
Поэтому, даже имея на руках различные карты, 3D-художникам приходится прикладывать усилия, чтобы текстура выглядела реалистично.
Чтобы получить реалистичный материал, специалисты по 3D используют черно-белое изображение текстуры для создания специальной карты черно-белого рельефа. Все белые пятна и линии на этой карте означают выпуклость, а черные — впадины. Используя карту рельефа в качестве основы, 3D-художники могут настроить выпуклость текстуры в программном обеспечении для 3D-графики, чтобы сделать ее более или менее рельефной и рельефной.
С помощью программного обеспечения для 3D художники могут изменять не только рельеф, но и прозрачность и отражение материала. Лучшим примером того, как все эти характеристики одинаково важны для фотореализма, является кожа. Нанесение на карту материала кожи — процесс, требующий больших затрат — он должен иметь точный рельеф, плотность и отражения одновременно. Но не волнуйтесь, работая с профессионалами, которые достаточно квалифицированы и опытны, даже самые сложные и подробные материалы выглядят на 100% реальными.
На первый взгляд создание 3D-модели выглядит как простая последовательность шагов, за которой легко следовать. Но из-за какой-либо ошибки проект может застрять на одном из этапов, а то и вернуться к предыдущим. Поэтому дальновидные 3D-специалисты всегда уделяют должное внимание всем этапам работы. Грамотно уточняют задачу, тщательно выстраивают геометрию и грамотно создают материалы. Осознавая все шаги, они могут избежать ошибок рендеринга и предоставить клиенту модель 100% качества.
Заинтересованы в создании первоклассных 3D-моделей? Выберите наши услуги 3D-моделирования и получите точные 3D-объекты фотореалистичного качества!
Создание 3D-моделей с помощью Autodesk Fusion 360
Об этом курсе
14 402 недавних просмотров
Дизайн — это первый этап цифрового производственного процесса. В этом курсе с помощью серии лекций и практических занятий мы рассмотрим подход дизайнера к процессу проектирования и производства — от концепции до 3D-модели. Мы начнем с применения проектного мышления для понимания потребностей пользователей, а затем изучим критерии проектирования по мере углубления в возможности создания эскизов, моделирования, визуализации и документирования в Autodesk® Fusion 360™.
Мы начнем с применения проектного мышления для понимания потребностей пользователей, а затем изучим критерии проектирования по мере углубления в возможности создания эскизов, моделирования, визуализации и документирования в Autodesk® Fusion 360™.
Гибкие сроки
Переустановите сроки в соответствии с вашим графиком.
Совместно используемый сертификатСовместно используемый сертификат
Получите сертификат по завершении
100% онлайн100% онлайн
Начните немедленно и учитесь по собственному графику.
СпециализацияКурс 3 из 5 в специализации
САПР и цифровое производство
Продвинутый уровеньПродвинутый уровень
Часов на прохождениеПрибл. 15 часов на выполнение
Доступные языкиАнглийский
Субтитры: арабский, французский, португальский (европейский), итальянский, вьетнамский, немецкий, русский, английский, испанский
Гибкие срокиГибкие сроки
Сбрасывайте сроки в соответствии с вашим графиком.
Общий сертификат
Получите сертификат по завершении
100% онлайн100% онлайн
Начните немедленно и учитесь по собственному графику.
СпециализацияКурс 3 из 5 в
Специализация САПР и цифровое производство
Продвинутый уровеньПродвинутый уровень
Часов, чтобы закончитьПрибл. 15 часов
Доступные языкиАнглийский
Субтитры: арабский, французский, португальский (европейский), итальянский, вьетнамский, немецкий, русский, английский, испанский
Инструктор
Autodesk
352 944 Учащиеся
22 КУРСЫ
Предлагаемые
AUTODESK
. Autodesk — мировой лидер в области технологий проектирования и производства. Обладая опытом в области архитектуры, проектирования, строительства, проектирования, производства и развлечений, мы помогаем новаторам во всем мире решать насущные проблемы сегодняшнего дня. Потому что мы верим, что если вы можете мечтать об этом, вы можете это сделать и в Autodesk.
Потому что мы верим, что если вы можете мечтать об этом, вы можете это сделать и в Autodesk.
Reviews
4.8
Filled StarFilled StarFilled StarFilled StarFilled Star159 reviews
5 stars
81.78%
4 stars
14.97%
3 stars
2.42%
2 звезды
0,13%
1 звезда
0,67%
ЛУЧШИЕ ОТЗЫВЫ ОТ СОЗДАНИЯ 3D-МОДЕЛЕЙ С AUTODESK FUSION 369
0002 by AKJun 2, 2020Во время курса я узнаю много нового о дизайне дронов и fusion 360. Это действительно помогает, если я хочу спроектировать свой собственный дрон с моими собственными возможностями.
Filled StarFilled StarFilled StarFilled StarFilled Starby NK12 ноября 2019 г.
Очень полезный курс, чтобы познакомиться с основами использования Autodesk Fusion 360, а также содержит много информации о том, как подойти к проблеме и найти правдоподобное решение.
от OGM 29 мая 2021 г.
Урок обучения навыкам проектирования для воспроизведения уникального дизайна 3D-модели на основе курса по созданию дрона-квадрокоптера.
Filled StarFilled StarFilled StarFilled StarStarот NMA 16 апреля 2020 г. Я научился проектировать корпус дрона. Но если бы курс мог рассказать больше о пространстве компонентов и дизайне для этого, это было бы действительно полезно.
Посмотреть все отзывы
О специализации CAD и цифровое производство
Будущее производства уже здесь, оно несет с собой радикальные изменения в способах проектирования, изготовления и использования вещей. И это разрушает каждую отрасль. Если вы обладаете необходимыми знаниями и инструментами, этот прорыв — это ваша возможность, независимо от того, являетесь ли вы предпринимателем, дизайнером или инженером.
Часто задаваемые вопросы
Когда я получу доступ к лекциям и заданиям?
Что я получу, подписавшись на эту специализацию?
Доступна ли финансовая помощь?
Есть ли у Autodesk дополнительные учебные ресурсы?
Как получить доступ к Autodesk Fusion 360 в качестве студента или преподавателя?
Как получить доступ к Fusion 360 в качестве профессионала?
Каковы системные требования для Fusion 360?
Как получить поддержку по установке Fusion 360?
Как опубликовать свои проекты в галерее Autodesk Fusion?
Как стать сертифицированным пользователем Autodesk?
Могу ли я получить доступ к этим учебным материалам бесплатно?
Есть вопросы? Посетите Справочный центр для учащихся.
Создание модели в Python | Документация
Когда вы создаете модель на Python, вы можете использовать ее с платформой Epicenter следующим образом:
- Создайте и загрузите код своей модели в папку Model вашего проекта.

- Понимание среды при запуске вашей модели Python в Epicenter.
- При необходимости создайте файл контекста модели и загрузите его в папку Model вашего проекта.
- При желании можно использовать пакет Epicenter в вашей модели. Этот пакет позволяет:
- Сохранение переменных в серверной базе данных Epicenter
- Зарегистрировать операции для автоматического выполнения
- Когда ваша модель будет завершена, вы можете создать пользовательский интерфейс для своего проекта. Вы можете:
- Доступ и обновление переменных
- Операции вызова
Создание и загрузка кода модели
Используйте пользовательский интерфейс Epicenter для загрузки кода модели (файлы .py) в папку Model в вашем проекте. Эта папка должна содержать все файлы вашей модели. Подпапки в порядке.
ВАЖНО : Имена файлов моделей (файлы .py) могут содержать буквы (A-Z, a-z), цифры (0-9) и знак подчеркивания (_). Однако имена файлов не могут содержать дефисы (-).
Однако имена файлов не могут содержать дефисы (-).
Понимание среды
Epicenter включает среду Python для запуска ваших моделей.
Версия Python
Версия Python по умолчанию — 2.7.12, независимо от того, используете ли вы версию API Epicenter v1 или v2. (Дополнительную информацию см. в истории версий API Epicenter.)
Вы можете изменить версию Python, включив файл контекста в папку Model вашего проекта. В частности, если вы установите «язык»: «python3» в вашем файле контекста, тогда версия Python будет 3.6.3.
Работа с пакетами и модулями
Epicenter по умолчанию включает следующие пакеты Python:
-
numpy -
scipy -
Эпицентр(относится к Эпицентру, подробности см. ниже)
Эти пакеты доступны независимо от того, используете ли вы Python 2.7.6 или 3.4.
Вы можете ссылаться на эти пакеты в своей модели, используя стандартные операторы from и import .
Чтобы использовать дополнительные пакеты Python в вашей модели:
- Загрузите исходный код в папку вашего проекта Model .
- Затем
импортируйтепо мере необходимости в определенные файлы модели.
Чтобы использовать пакет Epicenter в вашей локальной среде:
- Загрузите пакет Epicenter.py с сайта PiPY.
- Установите его (
pip install epicenter). - Используйте
import epicenterдля импорта установленного пакета.
Работа с подпапками
Когда вы помещаете код своей модели в папку Model , ваш основной файл — например, ваш основной модуль — должен находиться на верхнем уровне. Это файл, имя которого вы передаете API-интерфейсам Epicenter при создании новых прогонов.
Другие файлы могут находиться во вложенных папках или нет, в зависимости от того, как вы структурировали свою модель.
Использование пакета Epicenter
Пакет Epicenter, epicenter (Epicenter.py), позволяет сохранять переменные в серверной базе данных Epicenter, а также предоставляет другие утилиты.
Сохранение переменных
Когда вы работаете с платформой Epicenter, метаданные о каждом «запуске» — набор взаимодействий конечного пользователя с проектом и его моделью — автоматически сохраняются в серверной базе данных платформы Epicenter. Однако определенные переменные и их значения по умолчанию не сохраняются. (См. дополнительную информацию о Run Persistence.)
Несохраненные переменные доступны только в текущем сеансе. (Сеанс длится определенное время после того, как конечный пользователь перестал взаимодействовать с вашим проектом. Это время устанавливается в Model Session Timeout в настройках вашего проекта.)
Иногда это то поведение, которое вам нужно. : часто вам нужна ваша модель только для выполнения расчетов и анализа в текущем сеансе.
Для других симуляций или моделей вы хотите, чтобы конечные пользователи могли просмотреть часть или всю информацию из предыдущих запусков или возобновить работу с запуском, который они использовали ранее. В этих случаях вам необходимо сохранить переменные модели в серверной базе данных Epicenter.
Чтобы сохранить переменные из прогона в серверной базе данных платформы Epicenter из вашей модели Python:
В начале вашей модели Python импортируйте класс
Epicenterиз пакетаepicenter:из Эпицентра импорт Эпицентр
В вашей модели Python, если вы хотите сохранить переменную, добавьте строку:
Epicenter.record('имя_переменной_как_строка', фактическая_переменная)Например:
Epicenter.record('my_var_name', my_var_name)Этот вызов ставит переменные в очередь для сохранения из памяти в базу данных. Частота обработки очереди примерно каждые 30 секунд.
 Обычно этот вызов находится внутри функции Python в вашей модели, но это не обязательно.
Обычно этот вызов находится внутри функции Python в вашей модели, но это не обязательно.
Примечания:
Имена переменных Python не могут содержать пробелы.
После того как вы добавили вызов
record(), который сохраняет переменную, вам необходимо убедиться, что этот вызов выполняется в вашей модели. Например, если вызов находится внутри операции (функции), вы можете добавить операцию к компоненту (например, к кнопке) в своем пользовательском интерфейсе с помощью Flow.js. Или вы можете вызвать операцию напрямую.Используя
Epicenter.record, вы можете сохранять и обновлять стандартные типы переменных Python, используя Epicenter: логическое значение, число, строка, массив, словарь, объект.- Однако обратите внимание, что Epicenter хранит сложные типы переменных внутри как объекты JSON. В частности:
- логические значения Python хранятся как логические значения
- Строки Python хранятся как строки
- Числа Python хранятся в виде чисел (int, float и т.
 д.)
д.) - Массивы и кортежи Python хранятся в виде списков JSON
- Словари, объекты, типы (классы) и модули Python хранятся как объекты JSON
- Однако обратите внимание, что Epicenter хранит сложные типы переменных внутри как объекты JSON. В частности:
Важно : Вы не можете сохранить
nullв списках или словарях сEpicenter.record.
Регистрация операций для автоматического выполнения
При желании вы можете создать операции (функции) в своей модели, а затем зарегистрировать их, чтобы они автоматически вызывались при создании нового прогона.
Эта регистрация выполняется с помощью функции подписки в пакете epicenter (Epicenter.py).
Существует три возможности регистрации:
- Инициализация модели при создании нового прогона и
- Сброс модели при создании нового прогона на основе существующего прогона.
- Сразу же после восстановления запуска, если модель
restoreModeимеет значениеSNAPSHOT(подробности см. на странице Контекст модели 9).0359 режим восстановления ).
на странице Контекст модели 9).0359 режим восстановления ).
Например, в коде модели Python у вас будет:
# функция для инициализации модели может называться как угодно
определение инициализации модели():
...
# функция сброса модели, может называться как угодно
деф resetModel():
...
# функция, вызываемая после восстановления снимка, может называться как угодно
защита модели восстановления():
... Затем в файле основной модели (например, my_model.py ) вы регистрируете эти функции с помощью подписаться на функцию из класса Epicenter в пакете epicenter :
# в my_model.py
из эпицентра импорт Эпицентр
Epicenter.subscribe("инициализировать", initializeModel)
Epicenter.subscribe("сброс", resetModel)
Epicenter.subscribe("restore", restoreModel) Создание пользовательского интерфейса
Epicenter предоставляет несколько различных API-интерфейсов на разных уровнях абстракции, поэтому вы можете связать переменные модели и операции с вашим пользовательским интерфейсом. (Подробнее о различных вариантах для этого.)
(Подробнее о различных вариантах для этого.)
Когда ваша модель будет завершена, вы можете создать пользовательский интерфейс для своего проекта.
Доступ к переменным и их обновление
Хотя пример с сохранением переменных (выше) иллюстрирует базовую концепцию обращения к переменным, в большинстве реальных моделей используются объекты различных типов. Запись и доступ к переменным из списков, словарей, кортежей и других объектов включает в себя некоторые дополнительные тонкости.
Эпицентр хранит базовые и составные объекты внутри как объекты JSON.
Списки
Чтобы обновить или сослаться на весь список, используйте имя списка.
Чтобы обновить (исправить) или сослаться на элемент списка, вы можете использовать:
- числовые индексы с
[]
Например, учитывая список опубликованных_версий
опубликованных_версий = список( (0.8, 0.9, 1.0, 1.1, 1.2, 2.0, 2.1) )
вы можете сослаться на первый элемент, используя
publish_9versions[0.8, 0.9, 1.0, 1.1, 1.2, 2.0, 2.1))0459
Если вы используете Flow.js, это выглядит так:
И если вы используете Run API, это выглядит так:
curl -G \ 'https://api.forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/variables/published_versions[0]' \ --header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' curl -X ПАТЧ \ 'https://api.forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/переменные' \ --header 'Тип содержимого: приложение/json' \ --header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' \ --data '{"published_versions[0]": 0.65}'Использование
PATCHс индексами позволяет заменять определенные элементы в списке. Вы не можете добавлять новые элементы в список, просто используяPATCH. Например, попыткаPATCHнаpublish_versions[7]выдает ошибку, поскольку список содержит только элементы 0–6; вам нужно добавить к списку в вашей модели Python, прежде чем этот вызов API Epicenter будет работать.Использование
PATCHбез индексов позволяет заменить весь список.Важно : Обратите внимание, что вы не можете сохранить
nullв списках.Dicts
Чтобы сослаться на весь словарь, используйте имя словаря.
Чтобы обновить (исправить) или сослаться на элемент словаря, вы можете использовать:
- имена ключей, с
[]и строкой ключа
Например, учитывая state_info словарь
state_info = dict({ 'имя': 'Калифорния', 'аббревиатура': 'Калифорния',
'население': 38802500, 'допуск': 1850 }) вы можете сослаться на населения , используя
state_info['население']
Если вы используете Flow.js, это выглядит так:
И если вы используете Run API, это выглядит так:
завиток -G \
'https://api. forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/variables/state_info["население"]' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9'
curl -X ПАТЧ \
'https://api.forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/переменные' \
--header 'Тип содержимого: приложение/json' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' \
--data '{"state_info['население']": 3
forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/variables/state_info["население"]' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9'
curl -X ПАТЧ \
'https://api.forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/переменные' \
--header 'Тип содержимого: приложение/json' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' \
--data '{"state_info['население']": 3 00}'
Использование PATCH с именем ключа позволяет заменить дополнительные элементы в словаре. Приведенный выше запрос заменяет значение населения на . Если вместо этого вы использовали
--data '{"state_info": { "capital": "Sacramento" }}' , вы добавляете пар ключ:значение к state_info ; существующие пары ключ:значение сохраняются. Обратите внимание, что синтаксис PATCH для данных изменяется при замене значений вместо добавления новых пар ключ:значение.
Важно : Вы не можете полностью заменить словарь или сохранить null в словаре.
Кортежи
Чтобы обновить или сослаться на весь кортеж, используйте имя кортежа.
Чтобы сослаться на элемент кортежа, вы можете использовать:
- числовые индексы с
[]
Например, учитывая кортеж al_west
al_west = tuple(['дуб', 'лаа', 'море', 'хоу', 'текс'])
вы можете сослаться на первый элемент, используя
al_west[0]
Если вы используете Flow.js, это выглядит так:
И если вы используете Run API, это выглядит так:
curl -G \
'https://api.forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/variables/al_west[0]' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' Вы можете обновить весь кортеж, но не можете обновить только один элемент:
curl -X PATCH \
'https://api. forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/переменные' \
--header 'Тип содержимого: приложение/json' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' \
--data '{"al_west": ["окленд", "лос-анджелес", "сиэтл", "хьюстон", "техас"]}'
forio.com/v2/run/acme-simulations/sample-python-model/000001576501ce3b3387aa984aa94676ae17/переменные' \
--header 'Тип содержимого: приложение/json' \
--header 'Авторизация: носитель eyJhbGciOiJSUzI1NiJ9' \
--data '{"al_west": ["окленд", "лос-анджелес", "сиэтл", "хьюстон", "техас"]}' заменяет весь кортеж; но используя
--data '{"al_west[0]": "oakland"}' выдает ошибку.
Вызов операций
Вы можете вызвать любую операцию (функцию) в вашей модели Python из пользовательского интерфейса вашего проекта.
Например, в Flow.js добавьте операцию к элементу на странице или вызовите ее при создании цикла:
Создание модели - Sourcemodding
| Название / Назначение | Двигатель | |||
|---|---|---|---|---|
Source Studio Декомпилятор и компилятор Cannonfodders для исходников | ||||
Средство просмотра моделей Noesis Универсальное средство просмотра моделей | ||||
Sven Coop StudioMdl модернизированный studiomdl. | ||||
Преобразователь OBJ2SMD простой преобразователь, превращающий OBJ в SMD | ||||
Gmax Разбавленная бесплатная версия 3dsmax | ||||
Сообщество обновило HLMV Half-Life Model Viewer — совершенно новый инструмент, основанный на HLMV 1.25 и Jed's Model Viewer 1.36, который обеспечивает полностью перестроенное средство просмотра моделей. | ||||
Средство просмотра моделей Half-Life (исходный HLMV) Просмотр и настройка моделей перед их использованием в Engine | ||||
VTA Apply Это приложение позволяет применять анимацию VTA к файлам моделей SMD. | ||||
Плагины и инструменты 3dsmax Коллекция импортеров и экспортеров для 3dsmax | ||||
Плагины 3DS Max SMD и DMX Коллекция актуальных плагинов для 3dsmax и движков Valve | ||||
gssmt Мультитул GoldSource и Source (ранее известный как vmmt) со следующими функциями | ||||
Инструменты Blender Source Инструменты импорта и экспорта для Blender | ||||
gFlip и позволяет переворачивать отдельные smd анимации | ||||
Otis' Half-life Modelviewer Средство просмотра моделей, которое может открыть любую модель HL и просмотреть анимацию, масштабирование и т. | ||||
Valve Sketchup Tools Набор инструментов для Sketchup | ||||
Дополнение Valve Source для XSI Официальное дополнение для Softimage | ||||
zModeler Приложение для 3D-моделирования | ||||
3D-исследование Средство просмотра 3D-моделей, совместимое с моделями Half-Life | ||||
Blender3D SMD Exporter Инструменты экспорта для Blender | ||||
QC Mann помогает автоматизировать создание файлов QC | ||||
kHED Программа моделирования, простая для начинающих | ||||
Crowbar Декомпиляция, компиляция и просмотр моделей Source-Engine и GoldSrc в удобном интерфейсе | ||||
Редактор контроля качества — Compiler Advanced Edition Компилятор и редактор контроля качества | ||||
Средство просмотра моделей Jeds Half-Life (JHLMV) Улучшенное средство просмотра моделей для goldsrc | ||||
Modo в SMD Экспорт файлов Modo в формате SMD | ||||
MDL Gui GUI для компиляции и декомпиляции Goldsrc MDL. | ||||
Генератор моделей (MOG) Преобразование OBJ, BSP и VMF в OBJ или SMD | ||||
Декомпилятор MDL Декомпилировать файлы MDL обратно в файлы SMD | ||||
QME Это просмотрщик моделей для Quake 1. Когда-то платная программа стала бесплатной в 2006 году. | ||||
Jigglebone QCGen Помогает в процессе декомпиляции модели. | ||||
Half-Life EZ Weapon Creator Создает почти весь код, необходимый для добавления нового оружия в Half-Life | ||||
P2MV Средство просмотра моделей Xash4D | ||||
Набор инструментов для компиляции Hl2 Набор инструментов для компиляции моделей для игр Source | ||||
Инструменты WallWorm Инструменты Wallworm для экспорта и импорта в 3dsmax | ||||
Проппер Этот инструмент берет исходный файл карты (vmf) и создает файл . | ||||
Milkshape3D Пакет для 3D-моделирования со встроенной поддержкой двигателей Valves | ||||
SourceEyeCalc Изящный инструмент для расчета положения глаз модели | ||||
Kratisto's MDL Decompiler Декомпиляция игровых моделей | ||||
SMD экспортер Dvondrake для блендера Экспорт SMD из блендера | ||||
VMFtoSMD преобразование файлов карты VMF в файлы геометрии SMD | ||||
Компилятор Doggs MDL Графический интерфейс для studiomdl | ||||
MDLFlip и позволяет переворачивать отдельные smd анимации | ||||
MDL Texture Info Отображение информации о материалах, связанных с моделью | ||||
Polygon Cruncher Плагин 3D, уменьшающий количество полигонов в мешах | ||||
MDLstudio Это расширение кода mdl viewer из Half-Life SDK | ||||
Компилятор моделей Pretador - редактор контроля качества Просмотр полезной информации в файлах контроля качества | ||||
LithUnwrap Инструмент для бесплатной УФ-развертки | ||||
e_desc name=Rantis Extended Valve Tools Средство извлечения последовательности на основе Maya | ||||
Инструменты экспорта Maya от Prall Инструменты экспорта для Maya в Source | ||||
Преобразователь исходного кода в GoldSrc SMD Он разработан в качестве замены Milkshape в процессе переноса модели из Blender (с плагином SMD Tools) в моды на основе Half-Life 1 и Goldsrc. | ||||
Дерево простой в использовании генератор деревьев! | ||||
Source Model Finder Эта программа помогает находить модели для картографов Source. | ||||
Компилятор моделей NightFire Компилятор и декомпилятор моделей, специально разработанный для Nightfire | ||||
DoomMusics StudioMdl улучшенная StudioMdl | ||||
Studio Model Tool — Nightfire Плагин для NightFire Workshop | ||||
Left4Life StudioMDL модифицированный studimdl, компилирует очень большие модели | ||||
MDL Viewer Просмотр файлов модели для goldsrc | ||||
MESA Maya SMD Importer/Exporter | ||||
GUI Studio MDL Графический интерфейс для studiomdl | ||||
Плагины Lightwave Source Плагины движков Lightwave Source для импорта и экспорта | ||||
QCScript Сократите ежедневные трудности моделирования, правильно организовав свои модели и полностью контролируя их! | ||||
Ninja Ripper Копирование геометрии DirectX из игр в 3dsMax | ||||
Средство проверки модели Half-Life Код на C++, который поможет тем, кто хочет научиться экспортировать действительный меш Half-Life | ||||
Xash studioMDL Goldsrc Компилятор больших моделей Компилятор больших моделей для модов Half-Life от Xash-Team | ||||
IOgre Cinema4d to Source Плагины Cinema4D Source | ||||
Инструменты для работы с клапанами Небольшой набор инструментов для моделирования двигателей клапанов | ||||
Softimage/XSI Mod Tool Теперь снятый с производства инструмент Softimage когда-то был в авангарде создания 3D-контента. | ||||
Fragmotion Интерфейс 3D-моделирования для работы с моделями Half-Life | ||||
Модификатор модели (MOM) Изменение выбранных частей модели, таких как ссылки на текстуры |
Создание и анализ моделей на основе биохимических ограничений с использованием COBRA Toolbox v.3.0
- Обновление протокола
- Опубликовано:
- Laurent Heirendt 1 na1 ,
- Sylvain Arreckx 1 na1 ,
- Thomas Pfau
ORCID: orcid.
 org/0000-0001-5048-2923 2 ,
org/0000-0001-5048-2923 2 , - Себастьян Н. Мендоса ORCID: orcid.org/0000-0002-2192-5569 3,4 ,
- Энн Ришель 5 ,
- Альмут Хайнкен 1 ,
- Hulda S. Haraldsdóttir 1 ,
- Jacek Wachowiak 1 ,
- Sarah M. Keating 6 ,
- Vanja Vlasov 1 ,
- Stefania Magnusdóttir 1 ,
- Chiam Yu Ng 7 ,
- Немецкий преподобный 1 ,
- Alise žagare 1 ,
- Siu H. J. Chan 7 ,
- MARIK1592 1 ,
- Jennifer Modamio 1 ,
- John T. Sauls 8 ,
- Alberto Noronha 1 ,
- Aarash Bordbar 9 ,
- Benjamin Cousins 10 ,
- Diana C Эль Ассаль 1 ,
- Луис В. Валькарсель
ORCID: orcid.
 org/0000-0003-3769-5419 11 ,
org/0000-0003-3769-5419 11 , - Иньиго Апаоласа ORCID: orcid.org/0000-0002-8961-4513 11 ,
- Сьюзен Гадери 1 ,
- Masoud Ahookhosh 1 ,
- Marouen Ben Guebila 1 ,
- Andrejs Kostromins 12 ,
- Nicolas Sompairac 13 ,
- Hoai M. Le 1 ,
- Ding Ма 14 ,
- Юэкай Сун 15 ,
- Линь Ван 7 ,
- Джеймс Т. Юркович ORCID: orcid.org/0000-0002-9403-509X 16 ,
- Мигель А. П. Оливейра 1 ,
- Фан Т. Выонг 1 ,
- Леммер П. Эль Ассаль 1 ,
- Инна Куперштейн Orcid: orcid.org/0000-0001-8086-8915 13 ,
- Андреи Зиновьев 13 ,
- H. Scott Hinton 17 ,
- William A. A. A. Brry A. A. A. A. A. A. A. A.
 A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. Арагон Артачо 19 ,
A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. A. Арагон Артачо 19 , - Франсиско Х. Плейнс 11 ,
- Эгилс Сталидзанс 12 ,
- Alejandro Maass 3,4 ,
- Santosh Vempala 10 ,
- Michael Hucka 20 ,
- Michael A. Saunders 14 ,
- Michael A. Saunders 14 ,
- Michael A. Saunders 14 ,
- . Натан Э. Льюис ORCID: orcid.org/0000-0001-7700-3654 5,21 ,
- Томас Заутер 2 ,
- Бернхард Ø. Palsson 16,22 ,
- Инес Тиле 1 и
- …
- Ронан М. Т. Флеминг 1,23
Природные протоколы том 14 , страницы 639–702 (2019)Цитировать эту статью
14 тыс.
 обращений
обращений390 цитирований
33 Альтметрический
Сведения о показателях
Предметы
- Вычислительные модели
- Программное обеспечение
Abstract
Реконструкция и анализ на основе ограничений (COBRA) обеспечивает молекулярно-механистическую основу для комплексного анализа данных экспериментальной биологии молекулярных систем и количественного предсказания физико-химически и биохимически возможных фенотипических состояний. COBRA Toolbox — это комплексный набор программного обеспечения для настольных ПК с интероперабельными методами COBRA. Он нашел широкое применение в биологии, биомедицине и биотехнологии, поскольку его функции можно гибко комбинировать для реализации специализированных протоколов COBRA для любой биохимической сети. Этот протокол является обновлением для COBRA Toolbox v.1.0 и v.2.0. Версия 3.0 включает новые методы реконструкции с контролируемым качеством, моделирования, топологического анализа, планирования деформации и эксперимента, визуализации сети, а также сетевой интеграции хемоинформатических, метаболических, транскриптомных, протеомных и термохимических данных. Новая интеграция многоязычного кода также позволяет расширить область применения COBRA за счет высокоточных, высокопроизводительных и нелинейных решателей численной оптимизации для многомасштабного, многоклеточного и кинетического моделирования соответственно. Этот протокол предоставляет обзор всех этих новых функций и может быть адаптирован для создания и анализа моделей на основе ограничений в самых разных сценариях. COBRA Toolbox v.3.0 обеспечивает непревзойденную глубину методов COBRA.
Он нашел широкое применение в биологии, биомедицине и биотехнологии, поскольку его функции можно гибко комбинировать для реализации специализированных протоколов COBRA для любой биохимической сети. Этот протокол является обновлением для COBRA Toolbox v.1.0 и v.2.0. Версия 3.0 включает новые методы реконструкции с контролируемым качеством, моделирования, топологического анализа, планирования деформации и эксперимента, визуализации сети, а также сетевой интеграции хемоинформатических, метаболических, транскриптомных, протеомных и термохимических данных. Новая интеграция многоязычного кода также позволяет расширить область применения COBRA за счет высокоточных, высокопроизводительных и нелинейных решателей численной оптимизации для многомасштабного, многоклеточного и кинетического моделирования соответственно. Этот протокол предоставляет обзор всех этих новых функций и может быть адаптирован для создания и анализа моделей на основе ограничений в самых разных сценариях. COBRA Toolbox v.3.0 обеспечивает непревзойденную глубину методов COBRA.
Это предварительный просмотр содержимого подписки, доступ через ваше учреждение
Соответствующие статьи
Статьи открытого доступа со ссылками на эту статью.
Прогнозирование путей деградации фенольных соединений в микробиоте кишечника человека с помощью методов ферментативной неразборчивости
- Франческо Бальзерани
- , Даниэль Инохоса-Ногейра
- … Франсиско Дж. Плейнс
npj Системная биология и приложения Открытый доступ 12 июля 2022 г.
Модель метаболической сети Eriocheir sinensis icrab4665 в масштабе генома и анализ потребности в питании
- Цзинцзин Ли
- , Ифэй Гоу
- … Цзиншэн Сун
г.
 BMC Genomics Открытый доступ
28 июня 2022 г.
BMC Genomics Открытый доступ
28 июня 2022 г.Контроль окислительно-восстановительного потенциала для повышения ферментативности ферментативно гидролизованной кукурузной соломы, подвергнутой паровому взрыву, для производства бутанола
- Менглей Ся
- , Ди Ван
- … Мин Ван
Фабрики микробных клеток Открытый доступ 27 июня 2022 г.

Варианты доступа
Подписаться на журнал
Получить полный доступ к журналу на 1 год
99,00 €
всего 8,25 € за номер
Подписаться
Расчет налогов будет завершен во время оформления заказа.
Купить статью
Получите ограниченный по времени или полный доступ к статье на ReadCube.
$32,00
Купить
Все цены указаны без учета стоимости.
Рис. 1: Обзор ключевых концепций реконструкции и анализа на основе ограничений. Рис. 2: Непрерывная интеграция вновь разработанного кода выполняется на выделенном сервере с Jenkins. Рис. 3: Анализ баланса потока в нестационарном состоянии. Рис. 4. Пространства решений из установившихся потоков анизотропны, т. е. длинные в одних направлениях и короткие в других. Рис. 5. В процедуре OptForce ОБЯЗАТЕЛЬНЫЕ наборы определяются путем сопоставления диапазонов потоков, полученных с помощью анализа изменчивости потоков (FVA) штамма дикого типа (синие столбцы) и штамма с избыточной продуктивностью (красные столбцы). Рис. 6: Модель ветвления разработки COBRA Toolbox. Рис. 7 Рис. 8: Энергогенерирующий стехиометрически сбалансированный цикл. Рис. 9: Предсказанные методом OptForce воздействия на гиперпродукцию сукцината в E. coli (модель AntCore) в аэробных условиях. Рис. 10: Качественно прямые, количественно обратные реакции в многокомпонентной модели масштаба генома. Рис. 11: Визуализация метаболической сети человека.
е. длинные в одних направлениях и короткие в других. Рис. 5. В процедуре OptForce ОБЯЗАТЕЛЬНЫЕ наборы определяются путем сопоставления диапазонов потоков, полученных с помощью анализа изменчивости потоков (FVA) штамма дикого типа (синие столбцы) и штамма с избыточной продуктивностью (красные столбцы). Рис. 6: Модель ветвления разработки COBRA Toolbox. Рис. 7 Рис. 8: Энергогенерирующий стехиометрически сбалансированный цикл. Рис. 9: Предсказанные методом OptForce воздействия на гиперпродукцию сукцината в E. coli (модель AntCore) в аэробных условиях. Рис. 10: Качественно прямые, количественно обратные реакции в многокомпонентной модели масштаба генома. Рис. 11: Визуализация метаболической сети человека. Рис. 12: Выборочная визуализация модели ядра E. coli с помощью Paint4Net.
Рис. 12: Выборочная визуализация модели ядра E. coli с помощью Paint4Net. Каталожные номера
Palsson, B. Ø. Системная биология: реконструкция и анализ на основе ограничений (издательство Кембриджского университета, Кембридж, 2015).
Книга Google ученый
О’Брайен, Э. Дж., Монк, Дж. М. и Палссон, Б. О. Использование моделей в масштабе генома для прогнозирования биологических способностей. Cell 161 , 971–987 (2015).
ПабМед ПабМед Центральный Статья КАС Google ученый
Becker, S.A. et al. Количественное предсказание клеточного метаболизма с помощью моделей, основанных на ограничениях: набор инструментов COBRA. Нац. протокол 2 , 727–738 (2007).

КАС пабмед Статья Google ученый
Schellenberger, J. et al. Количественное предсказание клеточного метаболизма с помощью моделей, основанных на ограничениях: COBRA Toolbox v2.0. г. Нац. протокол 6 , 1290–1307 (2011).
Льюис, Н. Э., Нагараджан, Х. и Палссон, Б. О. Ограничение метаболических отношений генотип-фенотип с использованием методов филогении in silico. Нац. Преподобный Микробиол . 10 , 291–305 (2012).
Тиле, И. и Палссон, Б. Ø. Протокол для создания высококачественной метаболической реконструкции в масштабе генома. Нац. протокол 5 , 93–121 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Китано Х., Гош С. и Мацуока Ю. Социальная инженерия для виртуальной «большой науки» в системной биологии.
 Нац. хим. биол. 7 , 323–326 (2011).
Нац. хим. биол. 7 , 323–326 (2011).КАС пабмед Статья Google ученый
Бордбар А., Монк Дж. М., Кинг З. А. и Палссон Б. О. Модели на основе ограничений предсказывают метаболические и связанные с ними клеточные функции. г. Нац. Преподобный Жене. 15 , 107–120 (2014).
КАС пабмед Статья Google ученый
Майя, П., Роча, М. и Роча, И. Методы оптимизации деформации, основанные на ограничениях, in silico: поиск оптимальных клеточных фабрик. Микробиолог. Мол. биол. 80 , 45–67 (2016).
ПабМед Статья Google ученый
Hefzi, H. et al. Консенсусная реконструкция метаболизма клеток яичника китайского хомяка в масштабе генома. Клеточная система. 3 , 434–443.
 e8 (2016).
e8 (2016).КАС пабмед ПабМед Центральный Статья Google ученый
Юсуфи Ф.Н.К. и др. Биотехнология систем млекопитающих выявляет глобальные клеточные адаптации в рекомбинантной клеточной линии CHO. Клеточная система. 4 , 530–542.e6 (2017).
КАС пабмед Статья Google ученый
Zhuang, K. et al. Геномное динамическое моделирование конкуренции между Rhodoferax и Geobacter в бескислородных подземных средах. ISME J 5 , 305–316 (2011).
ПабМед Статья Google ученый
Джамшиди, Н. и Палссон, Б. Ø. Системная биология эритроцита человека. Клетки крови Мол. Дис. 36 , 239–247 (2006).
КАС пабмед Статья Google ученый
">Шломи Т., Кабили М. Н. и Руппин Э. Прогнозирование метаболических биомаркеров врожденных ошибок метаболизма человека. Мол. Сист. биол. 5 , 263 (2009).
ПабМед ПабМед Центральный Статья Google ученый
Саху, С., Францсон, Л., Йонссон, Дж. Дж. и Тиле, И. Сборник врожденных ошибок метаболизма, нанесенных на карту в метаболической сети человека. Мол. Биосист. 8 , 2545–2558 (2012).
КАС пабмед Статья Google ученый
Thiele, I. et al.
 Глобальная реконструкция метаболизма человека, управляемая сообществом. Нац. Биотехнолог. 31 , 419–425 (2013).
Глобальная реконструкция метаболизма человека, управляемая сообществом. Нац. Биотехнолог. 31 , 419–425 (2013).КАС пабмед Статья Google ученый
Пальярини, Р. и ди Бернардо, Д. Подход к моделированию в масштабе генома для изучения врожденных ошибок метаболизма печени: к пациенту in silico. г. Дж. Вычисл. биол. 20 , 383–397 (2013).
КАС пабмед ПабМед Центральный Статья Google ученый
Шакед И., Оберхардт М.А., Атиас Н., Шаран Р. и Руппин Э. Метаболическая сеть прогнозирования побочных эффектов лекарств. Клеточная система. 2 , 209–213 (2016).
КАС пабмед Статья Google ученый
Chang, R. L., Xie, L., Xie, L., Bourne, P. E. & Palsson, B. Нецелевые эффекты лекарств, предсказанные с использованием структурного анализа в контексте модели метаболической сети.
 PLoS-вычисление. Биол . 6 , e1000938 (2010 г.).
PLoS-вычисление. Биол . 6 , e1000938 (2010 г.).Келл, Д. Б. Системная биология, метаболическое моделирование и метаболомика в открытии и разработке лекарств. Препарат Дисков. Сегодня 11 , 1085–1092 (2006 г.).
КАС пабмед Статья Google ученый
Duarte, N.C. et al. Глобальная реконструкция метаболической сети человека на основе геномных и библиомических данных. Проц. Натл. акад. науч. США 104 , 1777–1782 (2007 г.).
КАС пабмед ПабМед Центральный Статья Google ученый
Swainston, N. et al. Recon 2.2: от реконструкции к модели метаболизма человека. Метаболомика 12 , 109 (2016).
ПабМед ПабМед Центральный Статья КАС Google ученый
">Zielinski, D.C. et al. Системно-биологический анализ факторов, лежащих в основе признаков метаболизма раковых клеток. науч. Респ. 7 , 41241 (2017).
КАС пабмед ПабМед Центральный Статья Google ученый
Мардиноглу, А. и др. Моделирование метаболизма гепатоцитов в масштабе генома выявило дефицит серина у пациентов с неалкогольной жировой болезнью печени. Нац. коммун. 5 , 3083 (2014).
ПабМед Статья КАС Google ученый
Karlstädt, A. et al. CardioNet: метаболическая сеть человека, подходящая для изучения метаболизма кардиомиоцитов. BMC Сист. биол. 6 , 114 (2012).

ПабМед ПабМед Центральный Статья КАС Google ученый
Gille, C. et al. HepatoNet1: комплексная метаболическая реконструкция гепатоцитов человека для анализа физиологии печени. Мол. Сист. биол. 6 , 411 (2010).
ПабМед ПабМед Центральный Статья КАС Google ученый
Мартинс Конде Пдо, Р., Заутер, Т. и Пфау, Т. Моделирование многоклеточных систем на основе ограничений. Фронт. Мол. Биоски . 3 , 3 (2016).
Бордбар, А. и др. Метаболическая сеть в масштабе генома с несколькими тканями для анализа физиологии систем всего организма. BMC Сист. биол. 5 , 180 (2011).
ПабМед ПабМед Центральный Статья Google ученый
Ицхак, К.
 и др. Моделирование метаболизма клеток на основе фенотипа выявляет метаболические нарушения рака. Элиф 3 , e03641 (2014).
и др. Моделирование метаболизма клеток на основе фенотипа выявляет метаболические нарушения рака. Элиф 3 , e03641 (2014).Артикул ПабМед Центральный Google ученый
Мардиноглу, А. и др. Интеграция клинических данных с моделью метаболизма адипоцитов человека в масштабе генома. Мол. Сист. биол. 9 , 649 (2013).
КАС пабмед ПабМед Центральный Статья Google ученый
Бордбар, А. и др. Персонализированные кинетические модели метаболизма цельных клеток для открытия в области геномики и фармакодинамики. Клеточная система. 1 , 283–292 (2015).
КАС пабмед Статья Google ученый
Shoaie, S. et al. Количественная оценка метаболических изменений микробиома кишечника человека, вызванных диетой.
 Клеточный метаб. 22 , 320–331 (2015).
Клеточный метаб. 22 , 320–331 (2015).КАС пабмед Статья Google ученый
Ногиец, К. Д. и Касиф, С. Дополнять или не дополнять: структура метаболической сети для пищевых добавок для человека. PLoS ONE 8 , e68751 (2013).
КАС пабмед ПабМед Центральный Статья Google ученый
Хайнкен А., Саху С., Флеминг Р. М. Т. и Тиле И. Характеристика метаболического симбиоза хозяин-микроб в кишечнике млекопитающих на системном уровне. Кишечные микробы 4 , 28–40 (2013).
ПабМед ПабМед Центральный Статья Google ученый
Heinken, A. et al. Функциональная метаболическая карта Faecalibacterium prausnitzii , полезного кишечного микроба человека. J. Бактериол. 196 , 3289–3302 (2014).

ПабМед ПабМед Центральный Статья КАС Google ученый
Magnúsdóttir, S. et al. Создание метаболических реконструкций в масштабе генома для 773 членов микробиоты кишечника человека. Нац. Биотехнолог. 35 , 81–89 (2017).
ПабМед Статья КАС Google ученый
Лакшманан М., Кох Г., Чанг Б.К.С. и Ли Д.-Ю. Программные приложения для анализа баланса потоков. Краткий биоинформ. 15 , 108–122 (2014).
ПабМед Статья Google ученый
Эбрахим А., Лерман Дж. А., Палссон Б. О. и Хайдук Д. Р. COBRApy: реконструкция и анализ на основе ограничений для Python. BMC Сист. биол. 7 , 74 (2013).
ПабМед ПабМед Центральный Статья Google ученый
">Heirendt, L., Thiele, I. & Fleming, R.M.T. DistributedFBA.jl: высокоуровневый высокопроизводительный анализ баланса потоков в Julia. Биоинформатика 33 , 1421–1423 (2017).
КАС пабмед ПабМед Центральный Google ученый
Латендресс, М., Крумменакер, М., Трупп, М. и Карп, П. Д. Построение и завершение моделей баланса потоков на основе баз данных путей. Биоинформатика 28 , 388–396 (2012).
КАС пабмед ПабМед Центральный Статья Google ученый
Karp, P.D. et al. Обновление Pathway Tools версии 19.0: программное обеспечение для информатики путей/генома и системной биологии. Краткий биоинформ. 17 , 877–890 (2016).

КАС пабмед Статья Google ученый
Сандве Г.К., Некрутенко А., Тейлор Дж. и Ховиг Э. Десять простых правил воспроизводимых вычислительных исследований. PLoS-вычисление. биол. 9 , e1003285 (2013 г.).
ПабМед ПабМед Центральный Статья Google ученый
Инс, округ Колумбия, Хаттон, Л. и Грэм-Камминг, Дж. Дело об открытых компьютерных программах. Природа 482 , 485–488 (2012).
КАС пабмед Статья Google ученый
Геворкян А., Бушелл М. Э., Авиньоне-Росса К. и Киржек А. М. СуррейFBA: инструмент командной строки и графический пользовательский интерфейс для моделирования на основе ограничений сетей метаболических реакций в масштабе генома. Биоинформатика 27 , 433–434 (2011).

КАС пабмед Статья Google ученый
Thorleifsson, S.G. & Thiele, I. rBioNet: расширение набора инструментов COBRA для реконструкции высококачественных биохимических сетей. Биоинформатика 27 , 2009–2010 (2011).
Солс, Дж. Т. и Буешер, Дж. М. Ассимиляция метаболических реконструкций в масштабе генома с помощью modelBorgifier. Биоинформатика 30 , 1036–1038 (2014).
КАС пабмед Статья Google ученый
Noronha, A. et al. ReconMap: интерактивная визуализация метаболизма человека. Биоинформатика 33 , 605–607 (2017).
КАС пабмед Google ученый
Gawron, P. et al. MINERVA — платформа для визуализации и курирования сетей молекулярного взаимодействия.
 нпдж Сист. биол. заявл. 2 , 16020 (2016).
нпдж Сист. биол. заявл. 2 , 16020 (2016).ПабМед ПабМед Центральный Статья КАС Google ученый
Оливье Б.Г., Ровер Дж.М. и Хофмейр Дж.-Х. S. Моделирование сотовых систем с помощью PySCeS. Биоинформатика 21 , 560–561 (2005).
КАС пабмед Статья Google ученый
Gelius-Dietrich, G., Desouki, A.A., Fritzemeier, C.J. & Lercher, M.J. Sybil — эффективное моделирование на основе ограничений в R. BMC Syst. биол. 7 , 125 (2013).
млн лет, Д. и др. Надежное и эффективное решение геномных моделей метаболизма и макромолекулярной экспрессии. г. Респ. 7 , 40863 (2017).
КАС пабмед ПабМед Центральный Статья Google ученый
">Кламт С. и фон Камп А. Интерфейс прикладного программирования для CellNetAnalyzer. Биосистемы 105 , 162–168 (2011).
КАС пабмед Статья Google ученый
Апаолаза И. и др. Подход in-silico для прогнозирования и использования синтетической летальности в метаболизме рака. Нац. Коммуна . 8 , 459 (2017).
Маранас, К. Д. и Зоморроди, А. Р. Методы оптимизации в метаболических сетях (Wiley, Нью-Йорк, 2016).
Книга Google ученый
">Thiele, I. et al. Многомасштабное моделирование метаболизма и синтеза макромолекул в г. E. coli и ее применение для эволюции использования кодонов. PLoS ONE 7 , e45635 (2012 г.).
КАС пабмед ПабМед Центральный Статья Google ученый
Feist, A.M. et al. Реконструкция метаболизма в масштабе генома для Escherichia coli K-12 MG1655, которая учитывает 1260 ORF и термодинамическую информацию. Мол. Сист. биол. 3 , 121 (2007).
ПабМед ПабМед Центральный Статья КАС Google ученый
">Ян Л. и др. Определение системной биологии основного протеома метаболизма и экспрессии согласуется с данными высокой пропускной способности. Проц. Натл. акад. науч. США 112 , 10810–10815 (2015).
КАС пабмед ПабМед Центральный Статья Google ученый
Борнстейн, Б. Дж., Китинг, С. М., Джоураку, А. и Хука, М. LibSBML: библиотека API для SBML. Биоинформатика 24 , 880–881 (2008).
КАС пабмед Статья Google ученый
">Brunk, E. et al. Recon 3D: ресурс, позволяющий получить трехмерное представление о вариациях генов в метаболизме человека. г. Нац. Биотехнолог . 36 , 272–281 (2018).
Ма, Д. и Сондерс, М. А. Решение многомасштабных линейных программ с использованием симплекс-метода с четырехкратной точностью. в Численный анализ и оптимизация , Vol. 134 (ред. Аль-Баали, М., Грандинетти, Л. и Пурнама, А.) 223–235 (Springer International Publishing, Чам, Швейцария, 2015 г.).
Флеминг, Р. М. Т. и Тиле, И. Элементарная кинетика с сохранением массы достаточна для существования неравновесной стационарной концентрации. г. Дж. Теор. биол. 314 , 173–181 (2012).
">Орт, Дж. Д., Тиле, И. и Палссон, Б. Ø. Что такое анализ баланса потоков? Нац. Биотехнолог. 28 , 245–248 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Фейст, А. М. и Палссон, Б. О. Целевая функция биомассы. Курс. мнение микробиол. 13 , 344–349 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Мелендес-Хевиа, Э. и Исидоро, А. Игра пентозофосфатного цикла. г. Дж. Теор. биол. 117 , 251–263 (1985).
ПабМед Статья Google ученый
">Yamada, T. et al. Прогнозирование и идентификация последовательностей, кодирующих орфанные ферменты, с использованием геномных и метагеномных соседей. мол. Сист. биол. 8 , 581 (2012).
ПабМед ПабМед Центральный Статья КАС Google ученый
Либерал Р. и Пинни Дж. В. Простые топологические свойства предсказывают функциональные неверные аннотации в метаболической сети. Биоинформатика 29 , i154–i161 (2013).
КАС пабмед ПабМед Центральный Статья Google ученый
Reed, J.
 L. et al. Системный подход к уточнению аннотации генома. Проц. Натл. акад. науч. США 103 , 17480–17484 (2006 г.).
L. et al. Системный подход к уточнению аннотации генома. Проц. Натл. акад. науч. США 103 , 17480–17484 (2006 г.).КАС пабмед ПабМед Центральный Статья Google ученый
Orth, J.D. & Palsson, B. Анализ заполнения пробелов реконструкции метаболической сети iJO1366 Escherichia coli для обнаружения метаболических функций. BMC Сист. биол. 6 , 30 (2012).
ПабМед ПабМед Центральный Статья Google ученый
Чанг, Р. Л. и др. Реконструкция метаболической сети Chlamydomonas дает представление об управляемом светом метаболизме водорослей. Мол. Сист. биол. 7 , 518 (2011).
ПабМед ПабМед Центральный Статья КАС Google ученый
Рольфссон, О., Палссон, Б.
 Ø. & Thiele, I. Реконструкция метаболизма человека Recon 1 направляет гипотезы о новых метаболических функциях человека. BMC Сист. биол. 5 , 155 (2011).
Ø. & Thiele, I. Реконструкция метаболизма человека Recon 1 направляет гипотезы о новых метаболических функциях человека. BMC Сист. биол. 5 , 155 (2011).ПабМед ПабМед Центральный Статья Google ученый
Рольфссон, О., Палья, Г., Магнусдоттир, М., Палссон, Б. Ø. & Thiele, I. Вывод метаболизма орфанных метаболитов человека из контекста их метаболической сети подтверждает активность глюконокиназы человека. г. Биохим. J. 449 , 427–435 (2013).
КАС пабмед Статья Google ученый
Сатиш Кумар, В., Дасика, М.С. и Маранас, К.Д. Автоматизированное курирование метаболических реконструкций на основе оптимизации. Биоинформатика BMC 8 , 212 (2007).
ПабМед ПабМед Центральный Статья КАС Google ученый
">Willemsen, A.M. et al. MetDFBA: включение измерений метаболомики с временным разрешением в анализ динамического баланса потоков. Мол. Биосист. 11 , 137–145 (2014).
ПабМед Статья КАС Google ученый
Клиссен, С., Ирганг, С., Кли, С., Джавалиско, П. и Николоски, З. Интеграция данных транскриптомики и метаболомики определяет метаболический ответ Chlamydomonas на лечение рапамицином. Plant J. 81 , 822–835 (2015).
КАС пабмед Статья Google ученый
Бордбар, А. и др. Выяснение динамической метаболической физиологии посредством сетевой интеграции количественной метаболомики во времени.
 г. Респ. 7 , 46249 (2017).
г. Респ. 7 , 46249 (2017).ПабМед ПабМед Центральный Статья Google ученый
Blazier, A.S. & Papin, J.A. Интеграция данных экспрессии в реконструкцию метаболической сети в масштабе генома. Фронт. Физиол . 3 , 299 (2012).
Opdam, S. et al. Систематическая оценка методов адаптации метаболических моделей в масштабе генома. Клеточная система. 4 , 318–329.e6 (2017).
КАС пабмед ПабМед Центральный Статья Google ученый
Эстевес, С. Р. и Николоски, З. Обобщенная структура для контекстно-зависимых методов извлечения метаболических моделей. Фронт. Растениевод . 5 , 491 (2014).
Влассис, Н., Пачеко, М. П. и Сотер, Т. Быстрая реконструкция компактных контекстно-зависимых моделей метаболических сетей.
 PLoS-компьютер. биол. 10 , e1003424 (2014).
PLoS-компьютер. биол. 10 , e1003424 (2014).ПабМед ПабМед Центральный Статья КАС Google ученый
Becker, S. A. & Palsson, B. O. Контекстно-зависимые метаболические сети согласуются с экспериментами. PLoS-вычисление. биол. 4 , e1000082 (2008 г.).
ПабМед ПабМед Центральный Статья КАС Google ученый
Зур Х., Руппин Э. и Шломи Т. iMAT: инструмент комплексного метаболического анализа. Биоинформатика 26 , 3140–3142 (2010).
КАС пабмед Статья Google ученый
Агрен, Р. и др. Реконструкция активных метаболических сетей в масштабе генома для 69 типов клеток человека и 16 типов рака с использованием INIT. Комп. PLoS. биол. 8 , e1002518 (2012 г.
 ).
).КАС Статья Google ученый
Джерби Л., Шломи Т. и Руппин Э. Вычислительная реконструкция тканеспецифических метаболических моделей: приложение к метаболизму печени человека. Мол. Сист. биол. 6 , 401 (2010).
ПабМед ПабМед Центральный Статья Google ученый
Ван Ю., Эдди Дж. А. и Прайс Н. Д. Реконструкция метаболических моделей в масштабе генома для 126 тканей человека с использованием mCADRE. BMC Сист. биол. 6 , 153 (2012).
ПабМед ПабМед Центральный Статья Google ученый
Кухар, М. Дж. Об использовании белкового обмена и периодов полураспада. Нейропсихофармакология 34 , 1172–1173 (2008).
ПабМед Статья КАС Google ученый
">Шустер, С. и Хильгетаг, К. Об элементарных режимах потока в системах биохимических реакций в установившемся режиме. J. Biol. Сист. 02 , 165–182 (1994).
Артикул Google ученый
Шиллинг, К. Х., Летчер, Д. и Палссон, Б. Ø. Теория системного определения метаболических путей и их использование для интерпретации метаболической функции с точки зрения путей. г. Дж. Теор. биол. 203 , 229–248 (2000).
КАС пабмед Статья Google ученый
Кламт, С. и др. От элементарных режимов потока к элементарным векторам потока: анализ метаболических путей с произвольными линейными ограничениями потока. PLoS-вычисление. биол. 13 , e1005409 (2017).

ПабМед ПабМед Центральный Статья КАС Google ученый
Бордбар, А. и др. Структура минимального метаболического пути согласуется с ассоциированными биомолекулярными взаимодействиями. Мол. Сист. биол. 10 , 737 (2014).
ПабМед ПабМед Центральный Статья КАС Google ученый
Гудмундссон С. и Тиле И. Эффективный в вычислительном отношении анализ изменчивости потока. BMC Bioinformatics 11 , 489 (2010).
ПабМед ПабМед Центральный Статья Google ученый
Haraldsdóttir, H. S., Cousins, B., Thiele, I., Fleming, R. M. T. & Vempala, S. CHRR: координировать наезд и бегство с округлением для единообразной выборки моделей на основе ограничений. Биоинформатика 33 , 1741–1743 (2017).

ПабМед ПабМед Центральный Статья Google ученый
Казинс, Б. и Вемпала, С. Гауссово охлаждение и алгоритмы для объема и гауссовского объема. СИАМ Дж. Вычисл. 47 , 1237–1273 (2018).
Казинс Б. и Вемпала С. Практический алгоритм объема. Матем. прог. Комп. 8 , 1–28 (2015).
Google ученый
Burgard, A.P., Pharkya, P. & Maranas, C.D. Optknock: двухуровневая структура программирования для определения стратегий нокаута генов для оптимизации микробного штамма. г. Биотехнология. биоинж. 84 , 647–657 (2003).
КАС пабмед Статья Google ученый
Патил, К.Р., Роча, И., Фёрстер, Дж. и Нильсен, Дж. Эволюционное программирование как платформа для метаболической инженерии in silico.
 BMC Bioinformatics 6 , 308 (2005).
BMC Bioinformatics 6 , 308 (2005).ПабМед ПабМед Центральный Статья КАС Google ученый
Лун, Д. С. и др. Крупномасштабная идентификация стратегий генетического дизайна с использованием локального поиска. Мол. Сист. биол. 5 , 296 (2009).
ПабМед ПабМед Центральный Статья Google ученый
Ранганатан С., Сазерс П. Ф. и Маранас С. Д. OptForce: процедура оптимизации для выявления всех генетических манипуляций, ведущих к целевому перепроизводству. PLoS-вычисление. биол. 6 , e1000744 (2010 г.).
ПабМед ПабМед Центральный Статья КАС Google ученый
Antoniewicz, M.R. et al. Анализ метаболических потоков в нестационарной системе: периодическая ферментация с подпиткой высокопродуктивного штамма E.
 coli , продуцирующего 1,3-пропандиол. Метаб. англ. 9 , 277–292 (2007).
coli , продуцирующего 1,3-пропандиол. Метаб. англ. 9 , 277–292 (2007).КАС пабмед ПабМед Центральный Статья Google ученый
Haraldsdóttir, H.S., Thiele, I. & Fleming, R.M.T. Сравнительная оценка программного обеспечения с открытым исходным кодом для сопоставления идентификаторов метаболитов в реконструкциях метаболических сетей: приложение к Recon 2. J. Cheminform. 6 , 2 (2014).
Preciat Gonzalez, G. A. et al. Сравнительная оценка алгоритмов картирования атомов для сбалансированных метаболических реакций: приложение к Recon 3D. Ж. Хеминформ. 9 , 39(2017).
ПабМед ПабМед Центральный Статья КАС Google ученый
Ким, С. и др. Базы данных веществ и соединений PubChem. Рез. нуклеиновых кислот. 44 , Д1202–Д1213 (2016).

КАС Статья пабмед Google ученый
Канехиса, М. и Гото, С. КЭГГ: Киотская энциклопедия генов и геномов. Рез. нуклеиновых кислот. 28 , 27–30 (2000).
Hastings, J. et al. Справочная база данных ChEBI и онтология для биологически значимой химии: улучшения на 2013 г. Nucleic Acids Res. 41 , Д456–Д463 (2013).
КАС пабмед Статья Google ученый
Sud, M. et al. LMSD: база данных структуры LIPID MAPS. Рез. нуклеиновых кислот. 35 , Д527–Д532 (2007 г.).
КАС пабмед Статья Google ученый
Форстер М., Пик А., Райтнер М., Шрайбер Ф. и Бранденбург Ф. Дж. Системная архитектура системы BioPath. In Silico Biol. 2 , 415–426 (2002).

КАС пабмед Google ученый
Уильямс, А. Дж., Ткаченко, В., Голотвин, С., Кидд, Р. и Макканн, Г. ChemSpider — создание основы для семантической сети путем размещения платформы базы данных по химии из краудсорсинга. г. Ж. Хеминформ. 2 , О16 (2010).
Wishart, D. S. et al. HMDB: база данных метаболизма человека. Рез. нуклеиновых кислот. 35 , Д521–Д526 (2007 г.).
КАС пабмед ПабМед Центральный Статья Google ученый
Рахман С.А. и др. Инструмент декодера реакций (RDT): извлечение признаков из химических реакций. Биоинформатика 32 , 2065–2066 (2016).
ПабМед ПабМед Центральный Статья КАС Google ученый
Кумар, А. и Маранас, К.
 Д. CLCA: максимальное количество общих запросов молекулярной подструктуры в базе данных MetRxn. J. Chem. Инф. Модель. 54 , 3417–3438 (2014).
Д. CLCA: максимальное количество общих запросов молекулярной подструктуры в базе данных MetRxn. J. Chem. Инф. Модель. 54 , 3417–3438 (2014).КАС пабмед Статья Google ученый
Симидзу Ю., Хаттори М., Гото С. и Канехиса М. Обобщенные модели реакций для предсказания неизвестных ферментативных реакций. Геном Информ. 20 , 149–158 (2008).
КАС пабмед Google ученый
Харальдсдоттир, Х. С. и Флеминг, Р. М. Т. Идентификация консервативных частей в метаболических сетях с помощью теоретико-графового анализа сетей атомных переходов. PLoS-вычисление. биол. 12 , e1004999 (2016).
ПабМед ПабМед Центральный Статья КАС Google ученый
Кламт, С., Хаус, У.-У. & Тайс, Ф.
 Гиперграфы и сотовые сети. PLoS-вычисление. Биол . 5 , e1000385 (2009 г.).
Гиперграфы и сотовые сети. PLoS-вычисление. Биол . 5 , e1000385 (2009 г.).Флеминг, Р. М. Т. и Тиле, И. фон Берталанффи 1.0: расширение набора инструментов COBRA для термодинамического ограничения метаболических моделей. Биоинформатика 27 , 142–143 (2011).
КАС пабмед Статья Google ученый
Fleming, R.M.T., Thiele, I. & Nasheuer, H.P. Количественное присвоение направленности реакции в моделях метаболизма, основанных на ограничениях: применение к Escherichia coli . Биофиз. хим. 145 , 47–56 (2009).
КАС пабмед ПабМед Центральный Статья Google ученый
Haraldsdóttir, H.S., Thiele, I. & Fleming, R.M.T. Количественное определение направленности реакции в многокомпонентной реконструкции метаболизма человека.
 г. Биофиз. J. 102 , 1703–1711 (2012).
г. Биофиз. J. 102 , 1703–1711 (2012).ПабМед ПабМед Центральный Статья КАС Google ученый
Нур, Э., Харальдсдоттир, Х.С., Майло, Р. и Флеминг, Р.М.Т. Непротиворечивая оценка энергии Гиббса с использованием компонентных вкладов. PLoS-вычисление. биол. 9 , e1003098 (2013 г.).
КАС пабмед ПабМед Центральный Статья Google ученый
Флеминг, Р. М. Т., Мэйс, К. М., Сондерс, М. А., Йе, Ю. и Палссон, Б. Ø. Вариационный принцип расчета неравновесных потоков и потенциалов в биохимических сетях масштаба генома. Ж. Теор. биол. 292 , 71–77 (2012).
КАС пабмед Статья Google ученый
Борода, Д. А., Лян, С.-Д. & Qian, H. Энергетический баланс для анализа сложных метаболических сетей.
 г. Биофиз. Дж . 83 , 79–86 (2002).
г. Биофиз. Дж . 83 , 79–86 (2002).Цянь, Х. и Берд, Д. А. Термодинамика стехиометрических биохимических сетей в живых системах, далеких от равновесия. Биофиз. хим. 114 , 213–220 (2005).
КАС пабмед Статья Google ученый
Флеминг, Р. М. Т., Тиле, И., Прован, Г. и Нашуер, Х. П. Комплексное стехиометрическое, термодинамическое и кинетическое моделирование стационарного метаболизма. г. Дж. Теор. биол. 264 , 683–692 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Шелленбергер, Дж., Льюис, Н. Э. и Палссон, Б. Ø. Устранение термодинамически невыполнимых петель в стационарных метаболических моделях. Биофиз. J. 100 , 544–553 (2011).
КАС пабмед ПабМед Центральный Статья Google ученый
">Флеминг, Р. М. Т., Влассис, Н., Тиле, И. и Сондерс, М. А. Условия двойственности между потоками и концентрациями в биохимических сетях. Ж. Теор. биол. 409 , 1–10 (2016).
КАС пабмед ПабМед Центральный Статья Google ученый
Арагон Артачо Ф.Дж., Флеминг Р.М.Т. и Вуонг П.Т. Ускорение алгоритма постоянного тока для гладких функций. Матем. Программа. 169 , 95–118 (2018).
Артачо, Ф. Дж. А. и Флеминг, Р. М. Т. Глобально сходящиеся алгоритмы поиска нулей дупломонотонных отображений. Оптим. лат. 9 , 1–16 (2014).
Google ученый
">Shannon, P. et al. Cytoscape: программная среда для интегрированных моделей сетей биомолекулярного взаимодействия. Рез. генома. 13 , 2498–2504 (2003).
КАС пабмед ПабМед Центральный Статья Google ученый
Кинг З.А. и др. Escher: веб-приложение для создания, обмена и встраивания богатых данными визуализаций биологических путей. PLoS-вычисление. биол. 11 , e1004321 (2015).
ПабМед ПабМед Центральный Статья КАС Google ученый
Куперштейн И. и соавт. NaviCell: веб-среда для навигации, курирования и обслуживания больших карт молекулярного взаимодействия.
 BMC Сист. биол. 7 , 100 (2013).
BMC Сист. биол. 7 , 100 (2013).ПабМед ПабМед Центральный Статья КАС Google ученый
Костромин А. и Сталидзанс Э. Paint4net: Расширение COBRA Toolbox для визуализации стехиометрических моделей метаболизма. Биосистемы 109 , 233–239 (2012).
КАС пабмед Статья Google ученый
Aurich, M.K. et al. Прогноз внутриклеточных метаболических состояний по данным внеклеточного метаболизма. Метаболомика 11 , 603–619 (2014).
ПабМед ПабМед Центральный Статья КАС Google ученый
Guebila, M.B. & Thiele, I. Оптимизация диеты на поздних стадиях лечения леводопой пациентов с болезнью Паркинсона. npj Сист. биол. заявл. 2 , 16013 (2016).
ПабМед ПабМед Центральный Статья КАС Google ученый
">Льюис, Н. Э. и др. Омические данные из эволюционировавшей E. coli согласуются с рассчитанным оптимальным ростом на основе моделей в масштабе генома. мол. Сист. биол. 6 , 390 (2010).
ПабМед ПабМед Центральный Статья КАС Google ученый
Тиле И., Флеминг Р. М. Т., Бордбар А., Шелленбергер Дж. и Палссон Б. Ø. Функциональная характеристика альтернативных оптимальных решений механизма транскрипции и трансляции Escherichia coli . Биофиз. J. 98 , 2072–2081 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Баллерстайн К.
 , фон Камп А., Кламт С. и Хаус У.-У. Минимальные наборы разрезов в метаболической сети являются элементарными модами в двойной сети. Биоинформатика 28 , 381–387 (2012).
, фон Камп А., Кламт С. и Хаус У.-У. Минимальные наборы разрезов в метаболической сети являются элементарными модами в двойной сети. Биоинформатика 28 , 381–387 (2012).КАС пабмед Статья Google ученый
фон Камп, А. и Кламт, С. Перечисление наименьших стратегий вмешательства в метаболические сети масштаба генома. PLoS-вычисление. биол. 10 , e1003378 (2014).
Артикул КАС Google ученый
Fujita, K.A. et al. Интеграция путей болезни Паркинсона в карту молекулярного взаимодействия. Мол. Нейробиол. 49 , 88–102 (2014).
КАС пабмед Статья Google ученый
Агрен, Р. и др. RAVEN Toolbox и его использование для создания метаболической модели в масштабе генома для г.
 Penicillium chrysogenum . PLoS-вычисление. биол. 9 , e1002980 (2013 г.).
Penicillium chrysogenum . PLoS-вычисление. биол. 9 , e1002980 (2013 г.).КАС пабмед ПабМед Центральный Статья Google ученый
Графахренд-Белау, Э., Клукас, К., Юнкер, Б.Х. и Шрайбер, Ф. FBA-SimVis: интерактивная визуализация метаболических моделей на основе ограничений. Биоинформатика 25 , 2755–2757 (2009).
КАС пабмед ПабМед Центральный Статья Google ученый
Rocha, I. et al. OptFlux: программная платформа с открытым исходным кодом для метаболической инженерии in silico. BMC Сист. биол. 4 , 45 (2010).
ПабМед ПабМед Центральный Статья КАС Google ученый
Poolman, MG ScrumPy: метаболическое моделирование с помощью Python. Сист. Биол .
 153 , 375–378 (2006).
153 , 375–378 (2006).Хоппе А., Хоффманн С., Гераш А., Гилле К. и Хольцюттер Х.-Г. FASIMU: гибкое программное обеспечение для серии вычислений баланса потоков в больших метаболических сетях. BMC Bioinformatics 12 , 28 (2011).
ПабМед ПабМед Центральный Статья Google ученый
Боэле, Дж., Оливье, Б.Г. и Теусинк, Б. FAME, анализ потоков и среда моделирования. BMC Сист. биол. 6 , 8 (2012).
ПабМед ПабМед Центральный Статья Google ученый
Ицхак, К., Габай, О., Коэн, Х. и Руппин, Э. Идентификация мишеней лекарственных средств, которые восстанавливают нарушенный метаболизм, и их применение к старению. Нац. коммун. 4 , 2632 (2013).
ПабМед Статья КАС Google ученый
Порнпуттапонг, Н., Нукаев, И. и Нильсен, Дж. Метаболический атлас человека: онлайн-ресурс по метаболизму человека. База данных 2015 , bav068 (2015).
Аркин А.П. и др. База знаний Министерства энергетики США по биологии. г. Нац. Биотехнолог. 36 , 566–569 (2018).
Кламт С., Саез-Родригес Дж. и Жиль Э. Д. Структурно-функциональный анализ сотовых сетей с помощью CellNetAnalyzer. BMC Сист. биол. 1 , 2 (2007).
ПабМед ПабМед Центральный Статья Google ученый
Чоудхури, А., Зоморроди, А. Р. и Маранас, К. Д. Методы двухуровневой оптимизации при расчете деформации. Комп. хим. англ. 72 , 363–372 (2015).
КАС Статья Google ученый
Тиле И., Джамшиди Н., Флеминг Р. М. Т. и Палссон Б. Ø. Реконструкция в масштабе генома механизма транскрипции и трансляции Escherichia coli : база знаний, ее математическая формулировка и ее функциональная характеристика. PLoS-вычисление. биол. 5 , e1000312 (2009 г.).
ПабМед ПабМед Центральный Статья КАС Google ученый
Аурих, М. К., Флеминг, Р. М. Т. и Тиле, И. MetaboTools: полный набор инструментов для анализа метаболических моделей в масштабе генома. Фронт. Физиол . 7 , 327 (2016).
Геворкян А., Пулман М. Г. и Фелл Д. А. Обнаружение стехиометрических несоответствий в биомолекулярных моделях. Биоинформатика 24 , 2245–2251 (2008).
КАС пабмед Статья Google ученый
Орт, Дж. Д. и Палссон, Б. Ø. Систематизация генерации недостающих метаболических знаний. Биотехнолог. биоинж. 107 , 403–412 (2010).
КАС пабмед ПабМед Центральный Статья Google ученый
Тиле И., Влассис Н. и Флеминг Р. М. Т. fastGapFill: эффективное заполнение пробелов в метаболических сетях. Биоинформатика 30 , 2529–2531 (2014).
Лайта, А. и Сильвестр, В. Справочник по нейрохимии и молекулярной нейробиологии г. (Спрингер, Бостон, 2008 г.).
Soh, KC & Hatzimanikatis, V. Сетевая термодинамика в постгеномную эпоху. Курс. мнение микробиол. 13 , 350–357 (2010).
КАС пабмед Статья Google ученый
Ахохош, М., Арагон, Ф.Дж., Флеминг, Р.М.Т. и Вуонг, П.Т. Локальная сходимость методов Левенберга-Марквардта при метрической субрегулярности Гельдера. Препринт на https://arxiv.org/abs/1703.07461 (2017).
Сан, Ю., Флеминг, Р. М. Т., Тиле, И. и Сондерс, М. А. Надежный анализ баланса потоков многомасштабных биохимических реакционных сетей. BMC Bioinformatics 14 , 240 (2013).
Скачать ссылки
Благодарности
Группа воспроизводимых результатов исследований (R3), в частности, К. Трефуа и Ю. Ярош из Люксембургского центра системной биомедицины, выражает благодарность за помощь в настройке виртуальной машины и сервера Jenkins. Это исследование финансировалось Национальным центром передового опыта в области исследований (NCER) болезни Паркинсона, Министерством энергетики США, Управлением передовых научных компьютерных исследований и биологических и экологических исследований в рамках программы «Научные открытия с помощью передовых вычислений», грант № . DE-SC0010429. Этот проект также получил финансирование от Программы исследований и инноваций HORIZON 2020 Европейского Союза в рамках соглашения о гранте №. 668738 и гранты Люксембургского национального исследовательского фонда (FNR) ATTRACT (FNR/A12/01) и OPEN (FNR/O16/11402054). Н.Э.Л. был поддержан NIGMS (R35 GM119850) и Фондом Ново Нордиск (NNF10CC1016517). М.А.П.О. был поддержан грантом Люксембургского национального исследовательского фонда (FNR) AFR/6669348. А.Р. была поддержана премией Lilly Innovation Fellows Award. Ф.Дж.П. был поддержан министром экономики и конкурентоспособности Испании (BIO2016-77998-R) и Программа ELKARTEK правительства Басков (KK-2016/00026). Я. был поддержан преддокторским грантом правительства Басков (PRE_2016_2_0044). Б.Ш.П. был поддержан Фондом Ново Нордиск через Центр биоустойчивости при Техническом университете Дании (NNF10CC1016517).
DE-SC0010429. Этот проект также получил финансирование от Программы исследований и инноваций HORIZON 2020 Европейского Союза в рамках соглашения о гранте №. 668738 и гранты Люксембургского национального исследовательского фонда (FNR) ATTRACT (FNR/A12/01) и OPEN (FNR/O16/11402054). Н.Э.Л. был поддержан NIGMS (R35 GM119850) и Фондом Ново Нордиск (NNF10CC1016517). М.А.П.О. был поддержан грантом Люксембургского национального исследовательского фонда (FNR) AFR/6669348. А.Р. была поддержана премией Lilly Innovation Fellows Award. Ф.Дж.П. был поддержан министром экономики и конкурентоспособности Испании (BIO2016-77998-R) и Программа ELKARTEK правительства Басков (KK-2016/00026). Я. был поддержан преддокторским грантом правительства Басков (PRE_2016_2_0044). Б.Ш.П. был поддержан Фондом Ново Нордиск через Центр биоустойчивости при Техническом университете Дании (NNF10CC1016517).
Информация об авторе
Примечания автора
Эти авторы внесли равный вклад: Laurent Heirendt, Sylvain Arreckx.

Авторы и филиалы
Luxembourg Centre for Systems Biomedicine, University of Luxembourg, Belvaux, Luxembourg
Laurent Heirendt, Sylvain Arreckx, Almut Heinken, Hulda S. Haraldsdóttir, Jacek Wachowiak, Vanja Vlasov, Stefania Magnusdóttir, German Preciat, Alise Žagare, Maike K Аурих, Кэтрин М. Клэнси, Дженнифер Модамио, Альберто Норонья, Дайана К. Эль Ассаль, Сьюзан Гадери, Масуд Ахукхош, Маруэн Бен Гебила, Хоаи М. Ле, Мигель А. П. Оливейра, Фан Т. Выонг, Леммер П. Эль Ассаль, Инес Тиле и Ронан М. Т. Флеминг
Исследовательского отделения наук о науках, Университет Люксембурга, Белво, Люксембург
Томас Пфау и Томас Саутер
Центр регуляции генома (Fondap 150
, Университет Чили, Сантьяго, Чили. Алехандро Маасс
Математика, Центр математического моделирования Чилийского университета, Сантьяго, Чили
Себастьян Н. Мендоса и Алехандро Маасс
Кафедра педиатрии, Калифорнийский университет, Сан-Диего, Медицинский факультет, Ла-Хойя, Калифорния, США
Энн Ришель и Натан Э.
 Льюис
ЛьюисЕвропейская лаборатория молекулярной биологии, Европейский институт биоинформатики (EMBL-EBI), Хинкстон , Кембридж, Великобритания
Сара М. Китинг
Факультет химического машиностроения, Университет штата Пенсильвания, Государственный колледж, Пенсильвания, США
Чиам Ю Нг, Сиу Х. Дж. Чан, Лин Ван и Костас Д. Маранас
Факультет физики, программы биоинформатики и системной биологии Калифорнийского университета, Сан-Диего, Ла-Хойя, Калифорния, США
John T. Sauls
Sinopia Biosciences, Сан-Диего, Калифорния, США
Aarash Bordbar
Центр алгоритмов и случайностей, Школа компьютерных наук, Технологический институт Джорджии, Атланта, Джорджия, США
Бенджамин Казинс и Сантош Вемпала
Департамент биомедицинской инженерии и наук, TECNUN, Университет Наварры, , Испания
Луис В. Валькарсель, Иньиго Апаолаза и Франсиско Дж. Плейнс
Институт микробиологии и биотехнологии Латвийского университета, Рига, Латвия
Андрей Костромин и Эгилс Сталидзанс
- Университеты Кури 9002 Paris Tech, Inserm, U900, Париж, Франция
Николя Сомпайрак, Инна Куперштейн и Андрей Зиновьев
Факультет управленческих наук и инженерии, Стэнфордский университет, Стэнфорд, Калифорния, США
Дин Ма и Майкл А.
 Сондерс
СондерсСтатистический факультет Мичиганского университета, Анн-Арбор, Мичиган, США
Юэкай Сан
Биоинженерный факультет Калифорнийского университета, Сан-Диего, Ла-Хойя , США
Джеймс Т. Юркович и Бернхард О. Palsson
Исследовательский фонд Университета штата Юта, Норт-Логан, Юта, США
H. Scott Hinton
Центр интегративной системной биологии и биоинформатики, факультет наук о жизни, Имперский колледж Лондона, Лондон, Великобритания
Уильям А. Брайант
Факультет математики, Университет Аликанте, Аликанте, Испания
Франсиско Дж. Арагон Артачо
Факультет вычислительных и математических наук, Калифорнийский технологический институт, США 0 90, Пасадена
Michael Hucka
Центр биоустойчивости Novo Nordisk Foundation, Калифорнийский университет, Сан-Диего, Ла-Хойя, Калифорния, США
Nathan E. Lewis
Центр биоустойчивости Фонда Ново Нордиск, Датский технический университет, Кемиторвет, Люнгбю, Дания
Bernhard Ø.
 Palsson
PalssonОтдел системной биомедицины и фармакологии, Академический центр по наркотикам Лейден, факультет науки, Университет Лейдена, Лейден, Нидерланды
Ронан М. Т. Флеминг
Авторитетные.
Вы также можете искать этого автора в PubMed Google Академия
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 Haraldsdóttir
HaraldsdóttirПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 Aurich
AurichПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 El Assal
El AssalПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 Le
LeПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 El Assal
El AssalПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Академия
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
 Saunders
SaundersПросмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Посмотреть публикации автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Просмотр публикаций автора
Вы также можете искать этого автора в PubMed Google Scholar
Вклады
SA: непрерывная интеграция, проверка кода, opencobra. github.io/cobratoolbox, Jenkins, Documenter.py, changeCobraSolver, поддержка запросов на вытягивание, учебные пособия, тесты, координация, рукопись и initCobraToolbox. LH: непрерывная интеграция, проверка кода, fastFVA (новая версия, тестирование и интеграция), MATLAB.devTools, opencobra.github.io, учебные пособия, тесты, поддержка запросов на вытягивание, координация, рукопись, initCobraToolbox и поддержка форума. TP: интеграция ввода-вывода и транскриптомной интеграции, учебные пособия, обзоры учебных пособий, разделы рукописи по интеграции ввода-вывода и транскриптомной интеграции, поддержка форума, поддержка запросов на включение и проверка кода. SNM: разработка и обновление алгоритмов расчета деформации, интеграция GAMS и MATLAB, а также учебные пособия. AR: методы интеграции транскриптомных данных, учебные пособия, раздел рукописи, посвященный интеграции транскриптомных данных, RuMBA, pFBA, метаболические задачи и обзор учебных пособий. AH: вклад в код многовидового моделирования, обзор учебника и тестирование.
github.io/cobratoolbox, Jenkins, Documenter.py, changeCobraSolver, поддержка запросов на вытягивание, учебные пособия, тесты, координация, рукопись и initCobraToolbox. LH: непрерывная интеграция, проверка кода, fastFVA (новая версия, тестирование и интеграция), MATLAB.devTools, opencobra.github.io, учебные пособия, тесты, поддержка запросов на вытягивание, координация, рукопись, initCobraToolbox и поддержка форума. TP: интеграция ввода-вывода и транскриптомной интеграции, учебные пособия, обзоры учебных пособий, разделы рукописи по интеграции ввода-вывода и транскриптомной интеграции, поддержка форума, поддержка запросов на включение и проверка кода. SNM: разработка и обновление алгоритмов расчета деформации, интеграция GAMS и MATLAB, а также учебные пособия. AR: методы интеграции транскриптомных данных, учебные пособия, раздел рукописи, посвященный интеграции транскриптомных данных, RuMBA, pFBA, метаболические задачи и обзор учебных пособий. AH: вклад в код многовидового моделирования, обзор учебника и тестирование. Х.С. Харальдсдоттир: термодинамика, консервативная часть и методы отбора проб. JW: документация. S.M.K.: поддержка ввода-вывода SBML. В.В.: учебные пособия. SM: многовидовое моделирование, обзор учебника и тестирование. CYN: обзор кода дизайна штамма, обзор учебного пособия и рукопись (введение OptForce/biotech). GP: учебные пособия и хемоинформатика для структур метаболитов и данных картирования атомов. А.Ж.: метаболическая картография. SHJC: навигация по решениям, код многовидового моделирования и обзор учебника. МКА: интеграция метаболомных данных. CMC: учебные пособия и тестирование. JM: метаболическая картография и учебные пособия по визуализации метаболической сети человека. JTS: код modelBorgifier и руководство. A.N.: виртуальная метаболическая интероперабельность человека. А.Б.: Метод и учебник MinSpan, надзор за методом и учебным пособием uFBA. B.C.: Равномерная выборка CHRR. DCEA: учебные пособия. L.V.V.: учебные пособия и реализация генетической MCS. IA: учебные пособия и реализация генетической MCS.
Х.С. Харальдсдоттир: термодинамика, консервативная часть и методы отбора проб. JW: документация. S.M.K.: поддержка ввода-вывода SBML. В.В.: учебные пособия. SM: многовидовое моделирование, обзор учебника и тестирование. CYN: обзор кода дизайна штамма, обзор учебного пособия и рукопись (введение OptForce/biotech). GP: учебные пособия и хемоинформатика для структур метаболитов и данных картирования атомов. А.Ж.: метаболическая картография. SHJC: навигация по решениям, код многовидового моделирования и обзор учебника. МКА: интеграция метаболомных данных. CMC: учебные пособия и тестирование. JM: метаболическая картография и учебные пособия по визуализации метаболической сети человека. JTS: код modelBorgifier и руководство. A.N.: виртуальная метаболическая интероперабельность человека. А.Б.: Метод и учебник MinSpan, надзор за методом и учебным пособием uFBA. B.C.: Равномерная выборка CHRR. DCEA: учебные пособия. L.V.V.: учебные пособия и реализация генетической MCS. IA: учебные пособия и реализация генетической MCS. С.Г.: совместимость с CellNetAnalyzer. MA: адаптивный решатель Левенберга – Марквардта. MBG: обзоры учебных пособий. А.К.: Код Paint4Net и руководство. NS: разработка инструмента и учебного пособия по метаболической картографии. HML: решатель оптимизации кардинальности. DM: решатели с четверной точностью. Ю.С.: многомасштабная переформулировка FBA. LW: обзор кода конструкции штамма, обзор учебного пособия и рукопись (OptForce). JTY: метод и руководство uFBA. МАПО: учебник. PTV: адаптивные решатели Левенберга – Марквардта и форсированная разность решателя выпуклой оптимизации. LPEA: интеграция и документирование хемоинформационных данных. И.К.: разработка инструмента и учебника по метаболической картографии. AZ: разработка инструмента и учебного пособия по метаболической картографии. Х.С. Хинтон: Основные учебники E. coli . W.A.B.: уточнение кода. FJAA: решатель дупломонотонных уравнений, решатель выпуклой оптимизации с повышенной разностью и адаптивные решатели Левенберга – Марквардта.
С.Г.: совместимость с CellNetAnalyzer. MA: адаптивный решатель Левенберга – Марквардта. MBG: обзоры учебных пособий. А.К.: Код Paint4Net и руководство. NS: разработка инструмента и учебного пособия по метаболической картографии. HML: решатель оптимизации кардинальности. DM: решатели с четверной точностью. Ю.С.: многомасштабная переформулировка FBA. LW: обзор кода конструкции штамма, обзор учебного пособия и рукопись (OptForce). JTY: метод и руководство uFBA. МАПО: учебник. PTV: адаптивные решатели Левенберга – Марквардта и форсированная разность решателя выпуклой оптимизации. LPEA: интеграция и документирование хемоинформационных данных. И.К.: разработка инструмента и учебника по метаболической картографии. AZ: разработка инструмента и учебного пособия по метаболической картографии. Х.С. Хинтон: Основные учебники E. coli . W.A.B.: уточнение кода. FJAA: решатель дупломонотонных уравнений, решатель выпуклой оптимизации с повышенной разностью и адаптивные решатели Левенберга – Марквардта. FJP: академический надзор, учебные пособия и реализация генетической MCS. Э.С.: академический надзор, Paint4Net и туториал. А.М.: академический надзор. С.В.: академический надзор и единый алгоритм выборки ЧРР. MH: академический надзор и поддержка ввода-вывода SBML. MAS: академический контроль, решатели с четверной точностью, вычисления в нулевом пространстве и выпуклая оптимизация. CDM: академический контроль и алгоритмы расчета деформации. NEL: академический контроль и кодирование, интеграция транскриптомных данных, RuMBA, pFBA, метаболические задачи и обзор учебных материалов. Т.С.: академический надзор и алгоритм FASTCORE. B.Ø.P.: академический надзор и управление openCOBRA. ИТ: академический контроль, учебные пособия, вклад в код и рукопись. RMTF: концептуализация, ведущий разработчик, академический надзор, архитектура программного обеспечения, проверка кода, разреженная оптимизация, вычисление нулевого пространства, термодинамика, вариационная кинетика, fastGapFill, выборка, консервативные части, визуализация сети, поддержка форума, учебные пособия и рукопись.
FJP: академический надзор, учебные пособия и реализация генетической MCS. Э.С.: академический надзор, Paint4Net и туториал. А.М.: академический надзор. С.В.: академический надзор и единый алгоритм выборки ЧРР. MH: академический надзор и поддержка ввода-вывода SBML. MAS: академический контроль, решатели с четверной точностью, вычисления в нулевом пространстве и выпуклая оптимизация. CDM: академический контроль и алгоритмы расчета деформации. NEL: академический контроль и кодирование, интеграция транскриптомных данных, RuMBA, pFBA, метаболические задачи и обзор учебных материалов. Т.С.: академический надзор и алгоритм FASTCORE. B.Ø.P.: академический надзор и управление openCOBRA. ИТ: академический контроль, учебные пособия, вклад в код и рукопись. RMTF: концептуализация, ведущий разработчик, академический надзор, архитектура программного обеспечения, проверка кода, разреженная оптимизация, вычисление нулевого пространства, термодинамика, вариационная кинетика, fastGapFill, выборка, консервативные части, визуализация сети, поддержка форума, учебные пособия и рукопись.
Автор, ответственный за переписку
Переписка с Ронан М. Т. Флеминг.
Заявление об этике
Конкурирующие интересы
Авторы не заявляют об отсутствии конкурирующих интересов.
Дополнительная информация
Примечание издателя: Springer Nature остается нейтральной в отношении юрисдикционных претензий в опубликованных картах и принадлежности к организациям.
Связанные ссылки
Ключевые ссылки, использующие этот протокол
Schellenberger, J. et al. Нац. Протоколы 6 , 1290–1307 (2011 г.): https://www.nature.com/articles/nprot.2011.308
Becker, S.A. et al. Нац. Протоколы 2 , 727–738 (2007 г.): https://www.nature.com/articles/nprot.2007.99
Этот протокол является обновлением Nat. протокол 2 , 727–738 (2007 г.): https://doi.org/10.1038/nprot.2007.99 и Nat. протокол 6 , 1290–1307 (2011): https://doi. org/10.1038/protex.2011.234
org/10.1038/protex.2011.234
Дополнительная информация
Дополнительные данные 1
Дополнительное механизм 1
MATLAB BASICS
Дополнительное механизм 1
MATLAB BASICS
Дополнительное руководство 1
MATLAB OR. основы
Дополнительное руководство 3
Внесение вклада в COBRA Toolbox с помощью git
Права и разрешения
Перепечатка и разрешения
Об этой статье
Дополнительная литература
Модель экспрессии генов на основе линейного программирования (LPM-GEM) предсказывает источник углерода для Bacillus subtilis.
- Кулвади Танамит
- Франциска Херхольд
- Райнер Кениг
Биоинформатика BMC (2022)
Модель метаболической сети Eriocheir sinensis icrab4665 в масштабе генома и анализ потребности в питании
- Цзинцзин Ли
- Ифэй Гоу
- Цзиньшэн Сунь
BMC Genomics (2022)
Контроль окислительно-восстановительного потенциала для повышения ферментативности ферментативно гидролизованной кукурузной соломы, подвергнутой паровому взрыву, для производства бутанола
- Менглей Ся
- Ди Ван
- Мин Ван
Фабрики микробных клеток (2022)
Микробные сообщества образуют богатые внеклеточные метаболомы, которые способствуют метаболическим взаимодействиям и способствуют толерантности к лекарствам.

- Джейсон С. Л. Ю
- Клара Коррейя-Мело
- Маркус Ральсер
Природа Микробиология (2022)
Улучшение производства рекомбинантного белка дрожжами посредством моделирования в масштабе генома с использованием ограничений протеома
- Фейран Ли
- Ю Чен
- Йенс Нильсен
Nature Communications (2022)
Комментарии
Отправляя комментарий, вы соглашаетесь соблюдать наши Условия и Правила сообщества.

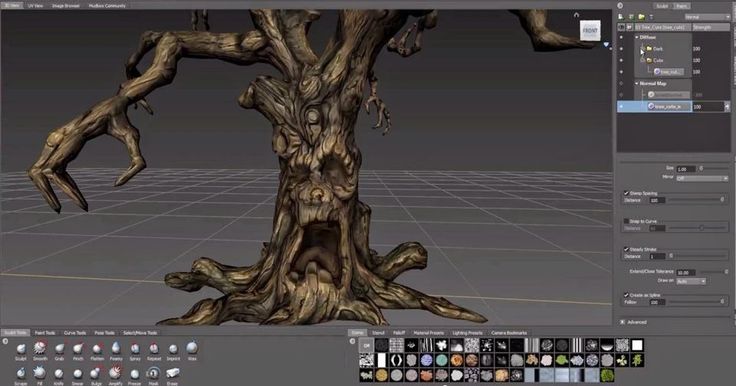 При этом начинающие свой путь новички зачастую не любят открывать видео “для нубов”, но и во второй категории мало что могут понять, заваливая комментарии вопросами, а после ещё и обижаясь на автора туториала.
При этом начинающие свой путь новички зачастую не любят открывать видео “для нубов”, но и во второй категории мало что могут понять, заваливая комментарии вопросами, а после ещё и обижаясь на автора туториала.
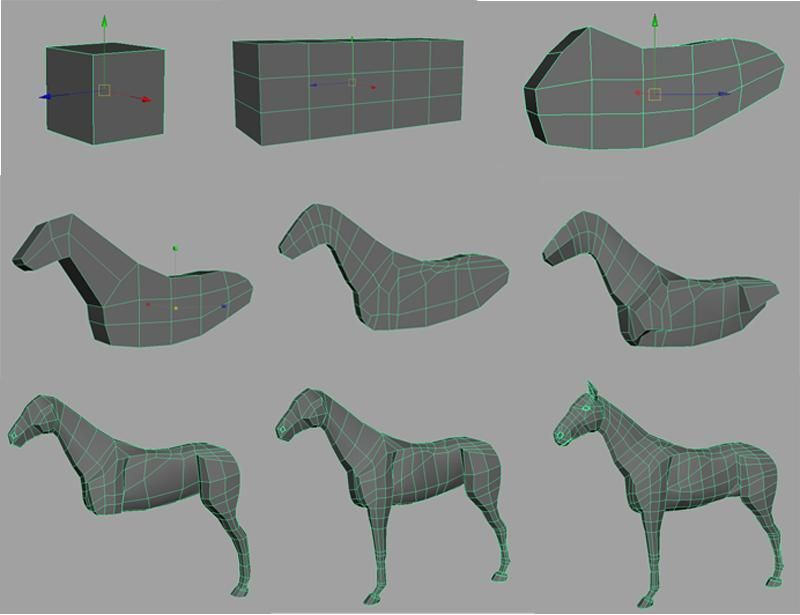
 На самом деле они не показывают, какие поля связаны друг с другом. Чтобы просмотреть ссылки, перейдите в раздел Power Pivot > Manage > Design > Relationships > Управление связями. В Excel можно перейти к разделу «>данных».
На самом деле они не показывают, какие поля связаны друг с другом. Чтобы просмотреть ссылки, перейдите в раздел Power Pivot > Manage > Design > Relationships > Управление связями. В Excel можно перейти к разделу «>данных».

